位于MC4R-CCBE1基因座的分子标记及其在猪背膘厚性状遗传改良中的应用
文献发布时间:2023-06-19 19:27:02
技术领域
本发明属于分子生物学技术领域,涉及一种位于MC4R-CCBE1基因座且影响猪背膘厚性状的分子标记及其在种猪遗传改良中的应用。
背景技术
随着人们生活水平的提高,消费者对猪肉的偏好由肥肉逐渐转变为瘦肉。在市场上,同一头猪的瘦肉价格普遍高于肥肉价格。由于猪瘦肉巨大的消费需求,全球育种目标已经从脂肪型猪向瘦肉型猪转变,提高瘦肉率是猪的主要育种目标。背膘厚与瘦肉率呈显著负相关关系,常被用来估算猪瘦肉率几十年来,育种工作者通过降低猪背膘厚来提高瘦肉率,使得背膘厚性状得到一定程度上的改良。但背膘厚是典型的数量性状,除了受多微效基因调控外,还受某些具有较大效应的基因(主效基因)调控。如若能利用分子遗传标记鉴定影响背膘厚性状的主效基因,并将其应用于猪背膘厚性状的遗传改良,将会加快猪背膘厚性状的遗传进展。因此,实有必要深入研究并确定影响猪背膘厚的主效QTL(quantitativetrait locus,QTL)及其主效基因,验证其对背膘厚性状的影响效应,最终建立高效准确的基因育种技术,将其应用于种猪肉质性状遗传改良中,从而改善猪肉品质。
全基因组关联分析(Genome-wide association study, GWAS)是解析猪数量性状的有效方法,而样本量和标记密度是影响GWAS结果的主要因素。由于背膘厚受到高强度人工选择,大效应单核苷酸多态(Single nucleotide polymorphisms, SNP)难以鉴定,因此,对于背膘厚的GWAS研究需要的样本量较大。目前,有关猪背膘厚等性状的GWAS研究,样本量一般在1000以内。此外,商业芯片的标记密度普遍偏低,重测序是增加标记密度有效方法,但重测序成本高。为控制降低成本,目前普遍采用重测序+基因型填充增加标记密度。即在小样本进行重测序,通过生信软件结合芯片数据和重测序数据对大样本进行基因型填充。
杜洛克猪常被用做杜长大商品猪(杜洛克猪×长白猪×大白猪)的终端父本。杜洛克作为终端父本直接影响杜长大的生产性能,而商品猪的生产性能直接影响到养殖场的经济效益。杜长大商品猪是我国市面上常见的商品猪之一。因此,对核心群杜洛克猪背膘厚性状进行改良,能够较大程度的将改良得到的优势遗传给商品猪后代,提高商品猪瘦肉率,从而提高养殖场的经济效益。
发明内容
为了解决现有技术中的不足,本发明通过对较大样本量的杜洛克种猪的背膘厚进行基于基因型填充数据的GWAS分析,鉴别到影响猪背膘厚的主效QTL和关键突变。在猪1号染色体上发现一个影响猪背膘厚的主效QTL,通过对GWAS最强关联位点与QTL区间内的显著SNP位点进行连锁不平衡分析,确定MC4R和CCBE1基因为潜在的主效基因。
本发明采用的具体方案如下:
本发明的第一方面是提供位于MC4R-CCBE1基因座的分子标记,所述分子标记的核苷酸序列如SEQ ID NO:1所示,其中序列中的M是T或C,导致猪背膘厚性状的差异。
本发明的第二方面是提供用于扩增上述分子标记的引物对,所述引物对包含primer-F和primer-R,其核苷酸序列如下:
上游引物primer-F:5’-TGGGCTTCCCTTGTCATGTA-3’;
下游引物primer-R:5’-TATGAATCAGGGGCACAAACCA-3’。
本发明的第三方面是提供一种试剂盒,所述试剂盒包含上述引物对。
本发明的第四方面是提供上述分子标记、引物对或试剂盒在如下任意一种中的应用:
a)、鉴定猪背膘厚性状;
b)、制备鉴定猪背膘厚性状的产品;
c)、猪遗传育种;
d)、制备提高猪背膘厚的产品。
本发明的第五方面是提供一种对猪背膘厚性状进行遗传改良的方法,使用序列表SEQ ID NO:1核苷酸序列,对待测猪的DNA进行PCR扩增,然后对扩增产物进行单核苷酸多态性检测,确定自序列表SEQ ID NO:1第278位碱基为T或者C,即该单核苷酸多态性位点对应于国际猪基因组11.1版本参考序列1号染色体上第161511936处的T>C突变,其中SEQ IDNO:1序列中的M是T或C,该位点位于CCBE1基因内,可导致猪背膘厚的差异。
本发明的第六方面是提供一种鉴定位于猪1号染色体上MC4R-CCBE1基因座且影响猪背膘厚性状的分子标记的基因型的方法,包含如下步骤:
(1)提取待测猪的基因组DNA;
(2)采用上述的引物对或试剂盒中的引物对对步骤(1)获得的基因组DNA进行PCR扩增,得到PCR扩增产物;
(3)对步骤(2)获得的PCR扩增产物进行测序,以便获得测序结果;
(4)基于所述测序结果,确定待测猪的位于猪1号染色体上MC4R-CCBE1基因座且影响猪背膘厚的SNP分子标记的基因型。
所述的猪优选为S21系杜洛克猪及其合成系。
有益效果:本发明利用杜洛克种猪,采用基因型填充技术获得猪全基因组序列数据,通过全基因组关联分析、连锁不平衡分析等方法,在猪1号染色体上发现一个影响猪背膘厚的主效QTL。通过对GWAS最强关联位点与QTL区间内的显著SNP位点进行连锁不平衡分析,确定MC4R和CCBE1基因为潜在的主效基因。更为重要的是,在猪达100kg体重时,最强关联位点g.1:161511936 T>C的TT型个体比CC型个体的背膘厚降低0.48mm,瘦肉率增加0.29%。在此基础上,构建一种高效且低成本的该突变位点的基因型的检测方法。通过对比研究g.1:161511936 T>C突变位点的不同基因型对猪背膘厚性状的影响,确定有利和不利的基因型,选择有利的基因型个体留种,淘汰不利的基因型个体,可以显著降低种群背膘厚度,从而达到改善猪背膘厚性状的目的。
附图说明
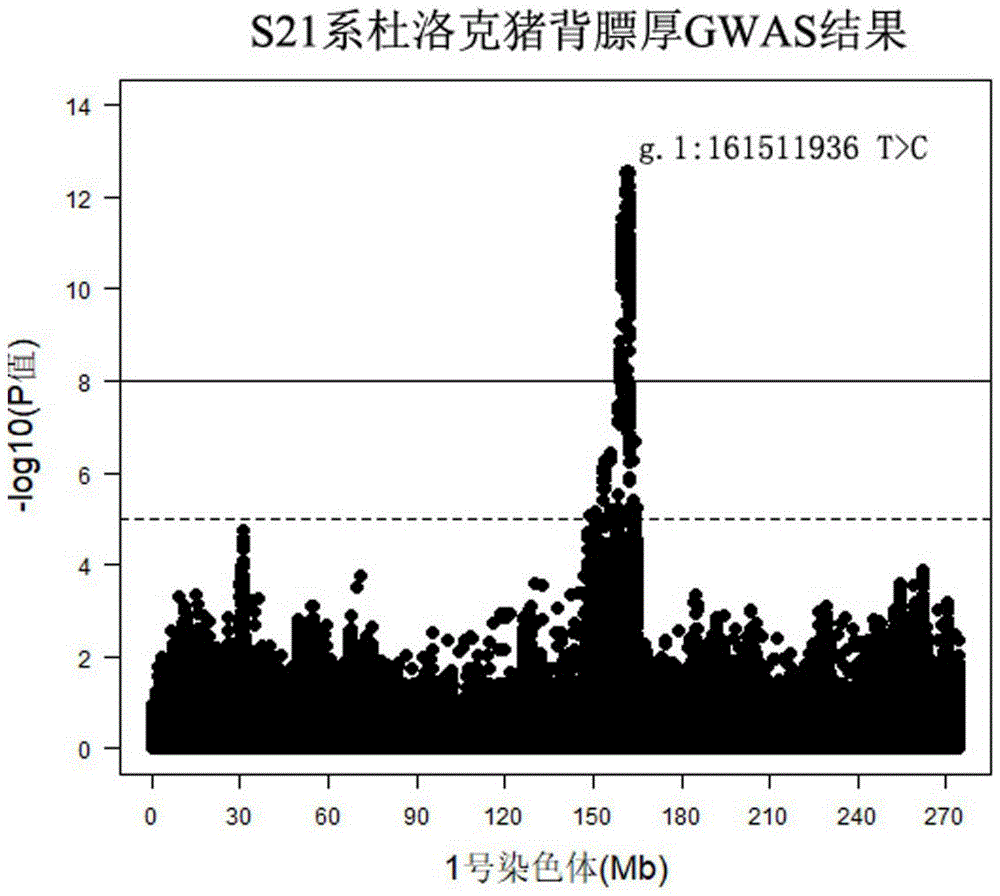
图1是S21系杜洛克在1号染色体上关于背膘厚性状的全基因组关联(GWAS)分析图。
图2是g.1:161511936 T>C位点不同基因型猪在100kg体重时的背膘厚结果分析图。
具体实施方式
为了深入研究可能影响猪背膘厚性状的主效QTL和基因,本发明利用杜洛克种猪,采用基因型填充技术获得猪全基因组序列数据,通过全基因组关联分析、连锁不平衡分析等方法,在猪1号染色体上发现一个影响猪背膘厚的主效QTL。通过对GWAS最强关联位点与QTL区间内的显著SNP位点进行连锁不平衡分析,确定MC4R和CCBE1基因为潜在的主效基因。结果显示,MC4R-CCBE1基因座(1:160772013-160774124;1:161320627-161556539)有78个SNP与最显著关联位点(g.1:161511936 T>C,
下面结合实施例及附图对本发明作进一步详细的描述,但本发明的实施方式不限于此。
实验猪群:本实验一共使用了3769头S21系杜洛克。
实施例1具体解释得到本发明中影响背膘厚的测定过程
在所有猪只体重达100kg左右时候,利用Aloka SSD 500V型活体B超测定仪测定所有试验猪只的背膘厚和眼肌厚,测定部位为第10~11肋间处。利用标配测量软件包计算背膘厚和眼肌厚,直接打印出影像及测量值。根据加拿大猪改良中心
本发明所使用的实验猪群群体为广东温氏食品集团股份有限公司种猪分公司的纯种S21系杜洛克3769头,为种猪分公司核心群,群体系谱记录详细。本实验共选取该资源群体中S21系杜洛克种猪,猪群在统一饲养标准下,自由采食、饮水,饲养至100±15kg体重。
实施例2具体解释得到本发明中基因标记的发明过程
(1)S21系杜洛克猪耳样组织DNA的提取方法参照标注苯酚-氯仿法提取全基因组DNA。用Nanodrop-ND1000分光光度计对纯种S21系杜洛克群体的DNA进行质量检测和浓度测定。A260/280比值在1.8~2.0,A260/230比值在1.7~1.9判定为合格。最后将合格的DNA样品统一稀释成50 ng/µl。
(2)猪全基因组50K SNP基因型检测:GeneSeek Genomic Profiler Porcine 50KSNP分型平台,采用Illumina Infinium的使用说明和标准流程进行芯片杂交与结果扫描。最后通过GenomeStudio软件读取基因型数据。用PLINKv1.90对获得的基因型数据进行质量控制,剔除检出率<99%,次等位基因频率(mimor allel frequency,MAF)<1%或偏离哈代温伯格平衡(Hardy-Weinberg Equilibrium,HWE)检验
(4)猪缺失基因型填充:从3769头S21系杜洛克猪中筛选155头猪进行重测序,使用Hiseq2500测序平台进行150bp的双端测序模式的高通量测序,获得155头S21系杜洛克猪重测序数据。将155个重测序个体组成参考群体,基于GATK软件构建S21系杜洛克猪重测序数据分析流程,获得参考群体的参考单倍型库。利用软件基于参考单倍型库将50K的SNPs芯片数据填充成全基因组测序数据。
(5)全基因组关联(GWAS)分析:为了消除群体层化效应,本发明通过GEMMA软件的线性混合模型单点回归分析对猪背膘厚进行基于填充数据的GWAS分析,分析模型中利用个体间基因组的相似度校正层化效应。对于基于基因型填充数据GWAS分析,染色体水平显著阈值和基因组水平显著阈值分别为1×10
GWAS分析结果如图1和附表1所示,最强关联位点g.1:161511936 T>C(
表1:位于MC4R-CCBE1基因座的SNP与最强关联位点g.1:161511936 T>C的关联程度(r2>0.80)统计表。
注:表中加粗的SNP ID为猪背膘厚最强关联位点,SNP位置对应于国际猪参考基因组11.1版本,r
(6)MC4R和CCBE1基因是潜在的影响猪背膘厚的主效基因
在上述2.64Mb处内包含了12个已注释基因,CDH20、MC4R、PMAIP1、CCBE1、LMAN1、CPLX4、RAX、GRP、SEC11C、ZNF532、MALT1和ALPK2基因。通过Plink v1.90软件计算最强关联位点g.1:161511936 T>C与上述2.64Mb处SNP的连锁不平衡程度r
(7)g.1:161511936 T>C的不同基因型与猪100kg体重时的背膘厚和瘦肉率表型的效应分析:根据表1可知,位于MC4R-CCBE1基因座的分子标记g.1:161511936T>C与背膘厚性状极显著相关(
表2:分子标记位点g.1:161511936 T>C(
实施例3具体解释发明检测SNP标记的发明过程
(1)含有与S21系杜洛克背膘厚显著相关的分子标记g.1:161511936 T>C的目的片段为1号染色体中的一段765bp(SEQ ID NO:1)的核苷酸序列。765bp的核苷酸序列扩增的上下游引物为primer-F和primer-R,其核苷酸序列如下:
上游引物primer-F:5’-TGGGCTTCCCTTGTCATGTA-3’;
下游引物primer-R:5’-TATGAATCAGGGGCACAAACCA-3’;
(2)PCR扩增的体系与条件设置
配置10μL体系,其中DNA样品1μL,上游引物0.3μL,下游引物0.3μL,PCR mix 5μL,ddH2O 3.4μL,PCR条件为95℃预变性5min,95℃变性30s,60℃退火30s,72℃延伸40s,共35个循环,最后延伸为72℃ 5min。
(3)DNA序列测序鉴定:序列测序在深圳华大基因科技有限公司进行,基因片段测正反两个反应。将所测得的序列与Ensembl基因组序列对比,得出对应SNP的突变。
SEQ ID NO:1的测序结果如下所示:
TGGGCTTCCCTTGTCATGTAACATAATTCATCTGGAGATATTGGGAAATTATGTTTAATGGGGAGACTCGACATTGCCACGTATTGTTCAGCTAGGTAAGTTACATATACGTATGTATTTCCTTTTGAAAAAAAAGATGTTGATGAAAGATTCAAAATAATCTATATTGAGTGGACCAGTGCAGGCAGGCCAGTGTATCCTTTGTTGGAGGTGGGGTTGGACTTAACTGTTTCACTCAAGTCTGCTTTGGGAGCATCCACTGTTTTTTATACAGAAG
注:序列中标注的M为突变位点,用标有下划线显示(括号中为突变碱基,为等位基因突变),在该序列的首尾加粗显示为引物序列结合位置。
实施例4分子标记位点(g.1:161511936 T>C)的效应分析
本发明提供一种位于猪1号染色体上的MC4R-CCBE1基因座且影响猪背膘厚的分子标记(g.1:161511936 T>C,
最后所应当说明的是,以上实施例仅用以说明本发明的技术方案而非对本发明保护范围的限制,尽管参照较佳实施例对本发明作了详细说明,本领域的普通技术人员应当理解,可以对本发明的技术方案进行修改或者等同替换,而不脱离本发明技术方案的实质和范围。
- 一种与长白猪背膘厚性状相关的SNP分子标记及其应用
- 猪背膘厚性状的分子标记及其应用
