针对消化道肿瘤的数据处理方法及系统
文献发布时间:2024-04-18 19:58:30
技术领域
本发明涉及医学数据分析领域,具体地涉及针对消化道肿瘤的数据处理方法。
背景技术
组织病理学切片包含重要的诊断、预后和生物学信息。数字病理学是指通过数字化技术将组织样本图像转换为数字格式的数据。
近年来,深度学习(deep learning,DL)方法在数字病理学领域展现出了巨大的潜力,为在消化道肿瘤诊断、预后预测和分子分类方面开发稳健的基于DL的预测模型提供了机会。目前,现有的数字病理数据存储平台(例如TCGA、TCIA等)虽然提供了大量的数字病理学数据、临床和分子测序数据,但这些平台存在标准化程度不高、数据来源单一、缺乏质控、标注处理等问题。这些问题会严重影响数字病理学数据在临床应用中的准确性和可靠性。此外,整理分子和预后标签信息需要专业的生物信息学和临床背景,这些对于计算学家来说是不擅长的。此外,这些平台缺乏用户友好的web接口,临床医生无法通过web界面轻松使用已经开发的深度学习模型来进行临床辅助诊断。因此,现有技术存在标准化、数据来源、质控、标注处理、分子和预后标签信息整理等方面的不足,同时缺少方便临床医生使用的用户友好的web接口。
数字病理学数据在现有技术中存在标准化程度不高、缺乏多样性、缺少质控和标注等环节处理等问题,同时分子和预后标签信息整理不完善。此外,缺乏用户友好的web接口使得临床医生难以使用已经开发的深度学习模型进行临床辅助诊断。这些问题的存在会影响数字病理学数据在深度学习模型中的应用效果和泛化能力,从而限制数字病理学数据在消化道肿瘤诊断、预后预测和分子分类等方面的应用。因此,需要提供一种新的技术来解决数字病理学数据在标准化、多样性、质控、标注处理、分子和预后标签信息整理以及用户友好的web接口等方面存在的问题。
发明内容
为解决现有技术中的至少部分技术问题,本发明提供针对消化道肿瘤的数据处理方法。具体地,本发明包括以下内容。
本发明的第一方面,提供一种针对消化道肿瘤的数据处理方法,包括:
获取消化道肿瘤相关的数据;
对消化道肿瘤相关的数据进行优化处理;
存储优化处理后的数据。
可选的,所述消化道肿瘤相关的数据,包括病理学数据、临床数据和/或分子测序数据,
对消化道肿瘤相关的数据进行优化处理,包括:
对获取的病理学数据进行肿瘤组织区域分割和颜色归一化处理,得到处理后的图像数据;
在处理后的图像数据中提取图像块特征,获取图像块特征的低维特征表示,从而得到病理学数据处理结果。
可选的,所述对获取的病理学数据进行肿瘤组织区域分割和颜色归一化处理的步骤之前,包括:
对病理学数据进行筛选和肿瘤组织区域注释,所述注释为可扩展标记语言格式。
可选的,所述对病理学数据进行筛选和肿瘤组织区域注释的步骤之后还包括:
对病理学数据进行质量控制,从而保证病理学数据的准确性和一致性。
可选的,所述对获取的病理学数据进行肿瘤组织区域分割,包括:
根据坐标点获取肿瘤组织区域轮廓C的最小外接矩阵M;
定义滑窗S对矩阵M以设定步长分割为图像块。
可选的,颜色归一化处理,包括:
将病理图像数据中的所有像素转换为相应的光密度(OD)值;
移除低于阈值β的光密度值,得到确定的光密度数据;
对确定的光密度数据进行奇异值分解(Singular Value Decomposition,SVD)得到奇异值向量和对应的特征向量;
由SVD分解获取的两个最大奇异值对应的两个特征向量形成一个平面;
将OD转换后的像素都投影到这个平面,并归一化为单位长度;
计算每个点相对与第一个SVD方向的角度,找到角度的鲁棒极值,最终将极值转换为光密度空间。
可选的,
所述在处理后的图像数据中获取图像块特征的低维特征表示,从而得到病理学数据处理结果,包括以下两种特征提取结果:
特征提取方法一:基于2D小波包变换构造多子图共生矩阵,提取基于多子图共生矩阵的纹理特征;
特征提取方法二:基于图像块使用神经网络计算图像块高语义低分辨率的特征表示。
可选的,所述基于图像块使用神经网络计算图像块高语义低分辨率的特征表示,包括:
使用在ImageNet上预训练的ResNet50模型,在网络的第三个残差块后使用自适应平均池化层将图像块转换为1024维特征向量。
可选的,临床数据进行优化处理,包括:
将临床数据定义为临床终点指标,临床终点指标包括总生存期、无进展生存期、无病生存期、疾病特异性生存期和复发转移的持续时间。
可选的,分子测序数据优化处理,包括:
计算肿瘤突变负荷,肿瘤突变负荷定义为全外显子序列中非同义突变的总数,将高肿瘤突变负荷阈值设置为样本人群中肿瘤突变负荷值的设定百分位数;
计算同源重组缺陷得分;HRD阳性的阈值设置为42。
本发明的第二方面,提供一种电子设备,所述电子设备包括:
至少一个处理器;以及,
与所述至少一个处理器通信连接的存储器;其中,
所述存储器存储有可被所述至少一个处理器执行的指令,所述指令被所述至少一个处理器执行,以使所述至少一个处理器能够执行第一方面任一所述的针对消化道肿瘤的数据处理方法。
可先的,所述电子设备进一步包括web接口,从而方便地供用户进行在线管理、存储和分析数据。
本发明的第三方面,提供一种计算机可读存储介质,该计算机可读存储介质存储计算机指令,该计算机指令用于使计算机执行第一方面任一所述的针对消化道肿瘤的数据处理方法。
本发明的第四方面,提供一种针对消化道肿瘤的数据处理系统,其用于执行根据第一方面所述的数据处理方法,所述系统包括:
a.数据采集模块,其设置为用于获取消化道肿瘤相关的数据;
b.处理模块,其设置为能够对消化道肿瘤相关的数据进行优化处理;
c.存储模块,其设置为存储优化处理后的数据。
本发明的针对消化道肿瘤的数据处理方法,通过对消化道肿瘤相关的数据进行优化处理,从而对数据进行标准化和统一化,便于做出更准确的诊断和预后预测。达到消化道肿瘤的数据标准化目的。
附图说明
图1为本实施例公开的针对消化道肿瘤的数据处理方法的流程图。
具体实施方式
现详细说明本发明的多种示例性实施方式,该详细说明不应认为是对本发明的限制,而应理解为是对本发明的某些方面、特性和实施方案的更详细的描述。
应理解本发明中所述的术语仅仅是为描述特别的实施方式,并非用于限制本发明。另外,对于本发明中的数值范围,应理解为具体公开了该范围的上限和下限以及它们之间的每个中间值。在任何陈述值或陈述范围内的中间值以及任何其他陈述值或在所述范围内的中间值之间的每个较小的范围也包括在本发明内。这些较小范围的上限和下限可独立地包括或排除在范围内。
除非另有说明,否则本文使用的所有技术和科学术语具有本发明所述领域的常规技术人员通常理解的相同含义。虽然本发明仅描述了优选的方法和材料,但是在本发明的实施或测试中也可以使用与本文所述相似或等同的任何方法和材料。本说明书中提到的所有文献通过引用并入,用以公开和描述与所述文献相关的方法和/或材料。在与任何并入的文献冲突时,以本说明书的内容为准。
本实施例公开了一种针对消化道肿瘤的数据处理方法,包括:
获取消化道肿瘤相关的数据;
对消化道肿瘤相关的数据进行优化处理;
存储优化处理后的数据。
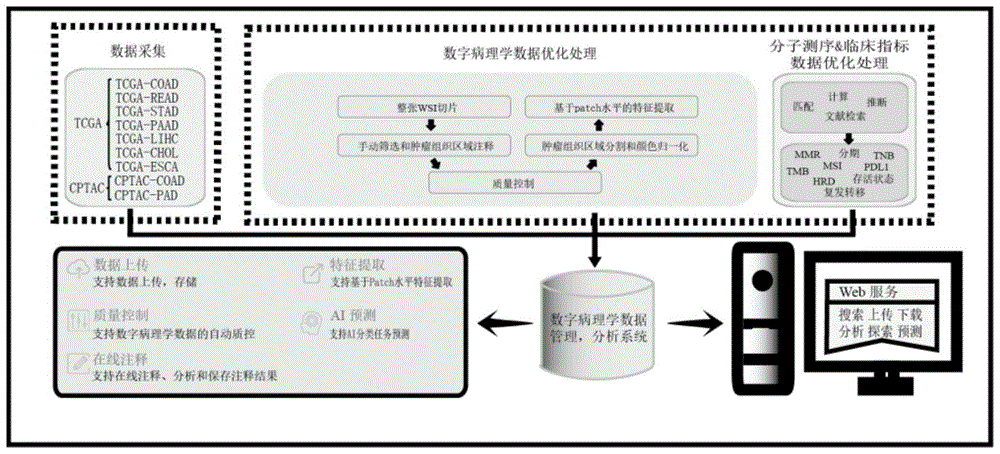
如图1所示,采集消化道肿瘤数字病理学数据,分子测序数据和临床数据;对数字病理学数据,分子测序数据和临床数据进行数据优化处理,以确保数据的准确性和可用性;将处理后的数字病理学数据存储到数据库中,以便后续的数据分析和处理;开发用户友好的web界面,提供研究者和临床医生进行数据的浏览、检索、图像显示、数据可视化和交互式分析等功能,方便用户进行深入分析。
数据采集:
本发明的数据采集包括从TCGA(The Cancer Genome Atlas)、CPTAC(ClinicalProteomic Tumor Analysis Consortium)在TCGA队列中,从https://portal.gdc.cancer.gov/repository/下载了原始FFPE切片、临床和基因测序数据。在CPTAC队列中,从https://www.cancerimagingarchive.net/下载了原始FFPE切片;从https://portal.gdc.cancer.gov/repository/下载了临床和基因测序数据。
数字病理学数据,临床数据,分子测序数据优化处理:
1.1、数字病理学数据优化处理方法:
本实施例的数字病理学数据优化处理流程包括数字病理学数据进行手动筛选和肿瘤组织区域注释,质量控制和特征提取等处理。通过以上优化处理,提高数字病理学数据在深度学习模型中的应用效果和泛化能力。具体而言,对原始数字病理学图像进行了以下优化处理:
1.1.1、数字病理学手动筛选和肿瘤组织区域注释:
首先,通过请专业的病理学家对所有数字病理学进行个体化手动审核,排除没有肿瘤组织或不属于消化道肿瘤(例如非侵袭性IPMNs、腺细胞癌、神经内分泌肿瘤等)的数字病理学切片,以确保所有输入图像均为高质量的肿瘤组织图像。为了进一步提高预测准确性,采用手动注释肿瘤区域的兴趣点的方法,具体而言,病理学家使用专业的数字病理学图像分析软件(如ASAP)对数字病理学图像进行手动注释,以标识肿瘤区域。并将注释导出为可扩展标记语言(XML)格式,包括X和Y坐标对应于注释区域。注释过程排除了坏死区域、血凝块、重叠区域、皱褶、原位癌或良性基质和上皮等区域,以确保标注的准确性和一致性。
1.1.2、质量控制:使用专业的数字病理学数据分析软件HistoQC对获得的数字病理学数据进行自动化质量控制。该软件可以生成质量控制指标,包括对颜色、亮度、对比度、清晰度等方面的评估,以确保数字病理学数据的准确性和一致性。
1.1.3、肿瘤组织区域分割和颜色归一化:
获得的数字病理学数据进行肿瘤组织区域分割和颜色归一化,具体实现方式如下。根据XML坐标点获取肿瘤组织区域轮廓C,
C={(x
的最小外接矩阵M。矩阵M的起始坐标点P,以及长和宽分别为:
P(x,y)=(min(x
M
wherex
定义滑窗S对矩阵M从左到右、从上到下以步长step,分割为小的图像块。whereS
为尽量减低不同批次间组织病理切片颜色差异,使用固定模板对肿瘤组织区域分割后的图像块(patch),进行颜色归一化:
(1)将所有颜色值转换为相对应的光密度(OD)值:
OD=-log
I是一个RGB颜色向量,并且每个分量都被归一化到[0,1]之间。
(2)移除低于阈值β的OD数据:
其中β定位为一个阈值,用于判断OD强度是否足够大。
(3)对确定好的OD进行SVD分解:
其中,OD为过滤后的光密度值,V和S分别为染色向量的矩阵和每个染色剂的饱和度。
(4)通过OD变换像素的SVD分解的两个最大奇异值对应的两个向量形成一个平面。
(5)将所有的OD转换后的向量都被投影到这个平面上,然后归一化为单位长度。
(6)计算每个点相对于第一个SVD向量的角度,找到角度的鲁棒极值,将极值转换回OD空间。
1.1.4、基于patch水平的特征提取:
采用了两种特征提取方法进行特征提取,具体而言,使用传统的颜色纹理特征提取方法(如灰度共生矩阵和灰度共生矢量等)和深度学习中常用的卷积神经网络(如ResNet和Inception等)进行特征提取。通过这些方法,可以得到高质量、低维度的特征向量,用于后续的分类、分割或其他图像分析任务。
(1)传统方法提取patch特征:
颜色和纹理是组织病理学图像分析中的重要特征。颜色矩是一种简单而有效的颜色描述符,如一阶矩(mean)、二阶矩(std)、三阶矩(var)等。由于颜色信息主要分布在不需要颜色空间量化和低特征维数的低阶矩中,因此选择了R、G和B通道的均值、方差和偏度来表征组织病理学图像的颜色特征。另外,使用基于2D小波包变换(waveletpackettransformation,WPT)创建的多子图共生矩阵(multi-sub-bands-co-occurrence matrix,WMCM)来提取病理切片图像的纹理特征。具体实现如下:
Step 1.构造WMCM
101、对patch执行一级2D-WPT,获得256x256像素的四张子图像:高频噪声子图像(High-High,HH),垂直细节子图像(Low-High,LH),水平细节子图像(High-Low,HL)和近似子图像(Low-Low,LL)。
102、计算整体细节子图像LHL
103、将LL和LHL量化到16Gy水平。将在LL和LHL中同时满足以下条件的像素点(s,t)定义为WMCM的元素m(i,j)。
LL(s,t)=i,LHL(s,t)=j,
104、标准化WMCM的m(i,j)
Step 2.提取基于WMCM的纹理特征:
让
201、Small Gray-level and Small Detail Advantage(SGSDA),定义为:
SGSDA测量了图像的亮度和纹理的厚度。
202、Small Gray-level and Big Detail Advantage(SGBDA),定义为:
SGBDA测量了图像的亮度和纹理的厚度。
203、Gray Level Average(GLA),定义为:
GLA测量了图像的整体亮度。
204、Detail Level Average(DLA),定义为:
DLA测量了纹理的整体厚度。
205、Gray Level Mean Square Error(GLMSE),定义为:
GLMSE测量了图像的灰度水平的离散度。
206、Detail Level Mean Square Error(DLMSE),定义为:
DLMSE测量了图像的纹理细节的离散度。
207、灰度值之间的线性相关性,定义为:
度量了WMCM在行或列方向上的相似度,并反应了图像的局部灰度相关性。
208、有序性,定义为:
有序性度量了纹理的规律性程度。
209、对比度,定义为:
对比度度量了WMCM的值分布和图像中的局部变化,并反应了图像的清晰度和纹理的深度。
210、反差矩,定义为:
反差矩度量了纹理在局部变化的程度,并反应了纹理的同质性。
211、熵,定义为:
熵反应了图像的灰度分布的复杂度。
(2)基于深度提取patch特征:
使用神经网络计算patch的低维特征表示。采用在ImageNet上预训练的ResNet50模型,在网络的第三个残差块后使用自适应平均池化层将图像块转换为1024维特征向量。
2、临床终点指标数据优化处理方法:
在本实施例中,系统地收集了患者的临床信息,包括诊断时间、治疗方案、治疗效果和随访情况等。基于上述信息对临床数据进行清洗、整合和统计等处理。定义了多个临床终点指标,包括总生存期、无进展生存期、无病生存期、疾病特异性生存期和复发转移的持续时间等。这些指标是衡量患者治疗效果和疾病进展的重要指标,可用于评估数字病理学图像分析的预测能力和临床应用价值。通过以上优化处理,可以得到准确、可靠的临床终点指标数据,为数字病理学图像分析结果的评估和临床应用提供可靠的依据。具体实施细节如下。
2.1、总生存期(Overall Survival,OS):对于每个患者,如果其生存状态为“死亡(Dead)”,则将其OS事件标记为1。如果患者的生存状态为“存活(Alive)”,则其OS事件标记为0。
2.2、无进展生存期(Progression-Free Interval,PFI):在本实施例中,定义了PFI用于评估患者治疗后的无进展生存时间。使用以下规则来确定PFI事件:
(1)如果患者出现了新的肿瘤事件,包括疾病进展、局部再发、远处转移、新的原发肿瘤或有肿瘤死亡,将该患者的PFI事件标记为1。
(2)如果患者没有出现新的肿瘤事件,则将该患者的PFI事件标记为0。
2.3、无病生存期(Disease-Free Interval,DFI):在本实施例中,定义了DFI用于评估患者治疗后的无病生存时间。使用以下规则来确定DFI事件:
(1)如果患者出现了新的肿瘤事件,无论是局部复发、远处转移还是新的原发肿瘤,包括类型为N/A的新肿瘤事件,将该患者的DFI事件标记为1。
(2)对于治疗结果字段treatment_outcome_first_course为“完全缓解/反应”的肿瘤类型,将其定义为无病状态,标记为0。如果该肿瘤类型没有treatment_outcome_first_course字段,则将residual_tumor字段中值为“R0”的患者定义为无病状态,标记为0。如果该肿瘤类型没有这两个字段,则将margin_status字段中值为“negative”的患者定义为无病状态,标记为0。
(3)如果该肿瘤类型没有上述任何字段,则将其DFI值定义为NA。
2.4、疾病特异性生存期(Disease-Specific Survival,DSS):定义了DSS用于评估患者治疗后的疾病特异性生存时间。使用以下规则来确定DSS事件:
(1)对于每个患者,如果其生存状态(Vital Status)为“死亡(Dead)”,且肿瘤状态(Tumor Status)为“有肿瘤(With Tumor)”,则将其DSS事件标记为1。
(2)如果患者死于与疾病相关的死因(Cause of Death),则其DSS状态也为1。
(3)如果患者的生存状态为“存活(Alive)”,或者生存状态为“死亡”但肿瘤状态为“无肿瘤(Tumor Free)”,则其DSS状态为0。
2.5、定义了复发或转移持续时间(Duration to Recurrence or Metastasis)作为一个临床终点指标,用于评估患者的疾病进程和治疗效果。将其定义为从手术时间开始到随访期间被诊断为复发或转移的时间。同时,制定了以下规则来确定复发转移标签:
(1)对于字段新肿瘤事件类型(new_tumor_event_type)为“复发”、“转移”或“复发|转移”,且字段初始治疗后新肿瘤事件发生的天数(days_to_new_tumor_event_after_initial_treatment)小于等于365(一年),则将该样本标记为一年内发生了复发转移,即state=1。
(2)对于字段初始治疗后新肿瘤事件(new_tumor_event_after_initial_treatment)为“NO”,且字段最后一次随访时间距离初始治疗时间的天数(days_to_last_followup)大于365(一年),则将该样本标记为一年内未发生复发转移,即state=0。
(3)对于同一个患者的多次记录中,将只保留其中一条记录,同时将其标签与该患者的其他记录一致。
(4)对于同一个患者的多次记录中,如果同时出现了0和1两种标签,将认为该患者在一年内发生了复发转移,即最终state=1。
3、分子测序数据优化处理方法
3.1、肿瘤突变负荷(Tumor mutation burden(TMB))被定义为全外显子序列中非同义突变的总数。TMB计算方法如下。
(1)使用“Maftools”R包计算全外显子序列中的非同义突变数量。表示为:Non-synonymous Mutations=计算全外显子序列中的非同义突变数量
(2)计算TMB。表示为:
TMB=Non-synonymous Mutations/全外显子序列的大小,其中,全外显子序列的大小按照40M计算。
(3)将高TMB阈值设置为样本人群中TMB值的第25个百分位数,
表示为:High TMB Threshold=样本人群中TMB值的第25个百分位数
3.2、在本实施例中使用“HRDetect”软件包计算了同源重组缺陷(HRD)得分,输入文件包括基因突变信息和拷贝数变异数据。HRD阳性的阈值设置为42。
4、Web门户使用HTML、CSS、JavaScript、Python、MySQL和Highcharts等技术实现,并提供了丰富的功能,如浏览、搜索、可视化和下载原始或标准化数据等。
因此,本实施例提供的消化道肿瘤的数字病理学数据处理、存储和自动化分析计算机程序产品和系统具有广泛的应用前景,可帮助研究人员和临床医生更好地理解消化道肿瘤的发病机制、预后预测和分子分类等方面,并为数字病理学数据在深度学习模型中的应用提供更准确、高质量的数据基础。
与现有技术相比,本实施例具有以下有益效果:
(1)全面的数字病理学图像数据集:该数据集涵盖了消化道肿瘤的5种主要类型,经过筛选的,提供了大量的标准化、手动注释、高质量的数字病理学数据,包括伴随患者诊断、预后和分子测序数据,能够为深度学习模型的开发和临床应用提供可靠的数据基础。
(2)多维分子和预后分类标签信息:本实施例的生物信息学家和临床医生团队基于分子和临床数据确认了超过50个分子和预后分类标签,提供了更加全面的数据信息,能够更好地支持深度学习方法在临床实践中的应用。
(3)自动化数字病理学图像分析和存储:本实施例的数字病理学数据处理和管理系统可以实现数字病理学图像的自动化分析和存储,包括数字病理学的自动化质控、特征提取和分类等。
(4)同时,提供了用户友好的界面,支持用户浏览、搜索、可视化和下载原始或标准化数据,从而提高数据的可访问性和可用性。
本实施例公开的电子设备包括存储器和处理器。该存储器用于存储非暂时性计算机可读指令。具体地,存储器可以包括一个或多个计算机程序产品,该计算机程序产品可以包括各种形式的计算机可读存储介质,例如易失性存储器和/或非易失性存储器。该易失性存储器例如可以包括随机存取存储器(RAM)和/或高速缓冲存储器(cache)等。该非易失性存储器例如可以包括只读存储器(ROM)、硬盘、闪存等。
该处理器可以是中央处理单元(CPU)或者具有数据处理能力和/或指令执行能力的其它形式的处理单元,并且可以控制电子设备中的其它组件以执行期望的功能。在本公开的一个实施例中,该处理器用于运行该存储器中存储的该计算机可读指令,使得该电子设备执行前述的本公开各实施例的多轨道InSAR的误差传递模型针对消化道肿瘤的数据处理方法全部或部分步骤。
本领域技术人员应能理解,为了解决如何获得良好用户体验效果的技术问题,本实施例中也可以包括诸如通信总线、接口等公知的结构,这些公知的结构也应包含在本公开的保护范围之内。
电子设备可以包括处理装置(例如中央处理器、图形处理器等),其可以根据存储在只读存储器(ROM)中的程序或者从存储装置加载到随机访问存储器(RAM)中的程序而执行各种适当的动作和处理。在RAM中,还存储有电子设备操作所需的各种程序和数据。处理装置、ROM以及RAM通过总线彼此相连。输入/输出(I/O)接口也连接至总线。
通常,以下装置可以连接至I/O接口:包括例如传感器或者视觉信息采集设备等的输入装置;包括例如显示屏等的输出装置;包括例如磁带、硬盘等的存储装置;以及通信装置。通信装置可以允许电子设备与其他设备(比如边缘计算设备)进行无线或有线通信以交换数据。但是应理解的是,并不要求实施或具备所有示出的装置。可以替代地实施或具备更多或更少的装置。
特别地,根据本公开的实施例,上文参考流程图描述的过程可以被实现为计算机软件程序。例如,本公开的实施例包括一种计算机程序产品,其包括承载在非暂态计算机可读介质上的计算机程序,该计算机程序包含用于执行流程图所示的方法的程序代码。在这样的实施例中,该计算机程序可以通过通信装置从网络上被下载和安装,或者从存储装置被安装,或者从ROM被安装。在该计算机程序被处理装置执行时,执行本公开实施例的多轨道InSAR的误差传递模型针对消化道肿瘤的数据处理方法的全部或部分步骤。
有关本实施例的详细说明可以参考前述各实施例中的相应说明,在此不再赘述。
根据本公开实施例的计算机可读存储介质,其上存储有非暂时性计算机可读指令。当该非暂时性计算机可读指令由处理器运行时,执行前述的本公开各实施例的多轨道InSAR的误差传递模型针对消化道肿瘤的数据处理方法的全部或部分步骤。
上述计算机可读存储介质包括但不限于:光存储介质(例如:CD-ROM和DVD)、磁光存储介质(例如:MO)、磁存储介质(例如:磁带或移动硬盘)、具有内置的可重写非易失性存储器的媒体(例如:存储卡)和具有内置ROM的媒体(例如:ROM盒)。
有关本实施例的详细说明可以参考前述各实施例中的相应说明,在此不再赘述。
以上结合具体实施例描述了本公开的基本原理,但是,需要指出的是,在本公开中提及的优点、优势、效果等仅是示例而非限制,不能认为这些优点、优势、效果等是本公开的各个实施例必须具备的。另外,上述公开的具体细节仅是为了示例的作用和便于理解的作用,而非限制,上述细节并不限制本公开为必须采用上述具体的细节来实现。
在本公开中,诸如第一和第二等之类的关系术语仅仅用来将一个实体或者操作与另一个实体或操作区分开来,而不一定要求或者暗示这些实体或操作之间存在任何这种实际的关系或者顺序,本公开中涉及的器件、装置、设备、系统的方框图仅作为例示性的例子并且不意图要求或暗示必须按照方框图示出的方式进行连接、布置、配置。如本领域技术人员将认识到的,可以按任意方式连接、布置、配置这些器件、装置、设备、系统。诸如“包括”、“包含”、“具有”等等的词语是开放性词汇,指“包括但不限于”,且可与其互换使用。这里所使用的词汇“或”和“和”指词汇“和/或”,且可与其互换使用,除非上下文明确指示不是如此。这里所使用的词汇“诸如”指词组“诸如但不限于”,且可与其互换使用。
另外,如在此使用的,在以“至少一个”开始的项的列举中使用的“或”指示分离的列举,以便例如“A、B或C的至少一个”的列举意味着A或B或C,或AB或AC或BC,或ABC(即A和B和C)。此外,措辞“示例的”不意味着描述的例子是优选的或者比其他例子更好。
还需要指出的是,在本公开的系统和方法中,各部件或各步骤是可以分解和/或重新组合的。这些分解和/或重新组合应视为本公开的等效方案。
可以不脱离由所附权利要求定义的教导的技术而进行对在此所述的技术的各种改变、替换和更改。此外,本公开的权利要求的范围不限于以上所述的处理、机器、制造、事件的组成、手段、方法和动作的具体方面。可以利用与在此所述的相应方面进行基本相同的功能或者实现基本相同的结果的当前存在的或者稍后要开发的处理、机器、制造、事件的组成、手段、方法或动作。因而,所附权利要求包括在其范围内的这样的处理、机器、制造、事件的组成、手段、方法或动作。
提供所公开的方面的以上描述以使本领域的任何技术人员能够做出或者使用本公开。对这些方面的各种修改对于本领域技术人员而言是非常显而易见的,并且在此定义的一般原理可以应用于其他方面而不脱离本公开的范围。因此,本公开不意图被限制到在此示出的方面,而是按照与在此公开的原理和新颖的特征一致的最宽范围。
为了例示和描述的目的已经给出了以上描述。此外,此描述不意图将本公开的实施例限制到在此公开的形式。尽管以上已经讨论了多个示例方面和实施例,但是本领域技术人员将认识到其某些变型、修改、改变、添加和子组合。
- 一种针对分布式数据库系统的数据处理方法、装置及系统
- 一种针对数据库的数据处理方法及装置,数据处理系统
