一种用于预测药物-靶标相互作用关系的方法和设备
文献发布时间:2023-06-19 09:32:16
技术领域
本发明涉及生物信息处理技术领域,特别涉及一种用于预测药物-靶标相互作用关系的方法和设备。
背景技术
众所周知,药物的开发是疾病治疗的重要前提,而药物-靶标关系的确认也是药物开发过程中的重要环节。尽管药物的开发在过去的几十年中取得了重要的进展,但是其财力和时间成本依然很高。随着系统生物学和网络药理学的发展,一个药物可以靶向多个不同的靶标,同样,一个靶标也能够被不同的药物所作用。在这个药物靶标关系网络中,药物靶标关系的确认能够加速药物的开发过程,了解药物的作用效果和疾病的治疗方案。
自08年提出网络药理学以来,以往单药物-靶向相互作用的思路被打破,而同年的论文《Yamanishi Yoshihiro,Araki Michihiro,Gutteridge Alex et al.Prediction ofdrug-target interaction networks from the integration of chemical and genomicspaces.[J].Bioinformatics,2008,24:i232-40》中描绘了并构建了酶、离子通道、G蛋白偶联受体(GPCR)和核受体的四类药物-靶标相互作用网络,其所提出方法的独创性在于将药物-靶标相互作用推论形式化为二分图的监督学习问题,该文中所使用的数据集也成为了基准标准集。而正是因为药物-靶标相互作用可以从定义上被抽象为计算机图论中的图并使用相关算法进行研究,使得计算机与药物或者生物学紧密相连,而在这基础上,围绕着以生物信息学为背景的多种研究方法在不同的立足点进行深层次的思考与发现。对于整个的药物-靶标相互作用网络来说,该网络被抽象为图论中的二部图。此时,药物和靶标分别作为二部图中的点集V,药物-靶标相互作用被抽象为边集E。故有模型G=(V,E)。附图1即为一个简单的二部图示例(其中星形节点为靶标,矩形节点为药物,其连接的边即为抽象后的药物-靶标相互作用)。
目前关于药物-靶标相互作用预测方法的研究还存在有以下缺陷:
1、使用的模型通常为具有药物-靶标相互作用的二部图模型,模型形式比较单一,其所能得到的药物-靶标相似度矩阵也较为简单,只能得到局部预测结果;
2、主流的生物方法主要依靠目标药物或靶标的全部信息,如完整的靶标3D结构等;同时带来的问题还有现有技术难以做大规模的筛选和预测,所得结果由于无针对性则往往花费较多而结果不尽如人意;
3、在面对越来越多的药物和靶标时还存在由于数量达到10^4~10^5量级而在中间计算过程中产生10^16以上存储空间需要,这样大的矩阵对于目前的计算水平是极其难以处理的。
发明内容
本发明旨在至少解决现有技术中存在的技术问题之一。为此,本发明提出一种用于预测药物-靶标相互作用关系的方法和设备。
本发明的第一方面,提供了一种用于预测药物-靶标相互作用关系的方法,其特征在于,包括以下步骤:
根据已知的药物-靶标相互作用关系及靶标-疾病相互作用关系构建药物-靶标-疾病的三层异构网络;
基于所述药物-靶标-疾病的三层异构网络构建药物相似度矩阵和靶标相似度矩阵,所述靶标相似度矩阵由靶标间高斯核相似度矩阵和靶标-疾病高斯核相似度矩阵拟合得到;
计算所述药物相似度矩阵和所述靶标相似度矩阵克罗内克积,并通过正则化最小二乘法得到预测结果;
对所述预测结果进行验证。
本发明的第二方面,提供了一种用于预测药物-靶标相互作用关系的设备,包括:至少一个控制处理器和用于与所述至少一个控制处理器通信连接的存储器;所述存储器存储有可被所述至少一个控制处理器执行的指令,所述指令被所述至少一个控制处理器执行,以使所述至少一个控制处理器能够执行如本发明第一方面所述的用于预测药物-靶标相互作用关系的方法。
本发明的第三方面,提供了一种计算机可读存储介质,其特征在于,所述计算机可读存储介质存储有计算机可执行指令,所述计算机可执行指令用于使计算机执行如本发明第一方面所述的用于预测药物-靶标相互作用关系的方法。
根据本发明的实施例,至少具有如下技术效果:
(1)引入靶标-疾病相互作用并结合药物-标靶相互作用构建了三层异构网络,基于三层异构网络构建药物相似度矩阵和靶标相似度矩阵,靶标相似度矩阵由靶标间高斯核相似度矩阵和靶标-疾病高斯核相似度矩阵拟合得到,对比传统的预测方法,采用了更为完整的网络结构模型,建立了更为复杂的相似度矩阵空间,从更多的角度预测了全新的药物-靶标相互作用。
(2)使用了克罗内克积的正则化最小二乘法预测最终结果,在计算过程中避免了超规模的矩阵运算。
(3)通过对本发明得到的预测结果进行验证,相较于常用的FLapRLS方法和RLS_Kron方法,有着更优的预测性能。本发明得到的且未被化学性验证的新的药物-靶标相互作用具有很高的研究价值,其能够使得后续的化学验证试验既可以有针对性的进行,避免大范围没有明确目标的重复性试验。同时因为给出的药物已经通过临床试验等步骤,在最终商业化的重新使用上避免了新研发药物的过长周期。
本发明的附加方面和优点将在下面的描述中部分给出,部分将从下面的描述中变得明显,或通过本发明的实践了解到。
附图说明
本发明的上述和/或附加的方面和优点从结合下面附图对实施例的描述中将变得明显和容易理解,其中:
图1为现有的二部图示例;
图2为本发明实施例提供的用于预测药物-靶标相互作用关系的方法的流程示意图;
图3为本发明实施例提供的用于预测药物-靶标相互作用关系的方法的流程示意图;
图4为本发明实施例提供的药物-靶标-疾病的三层异构网络的示意图;
图5为本发明实施例提供的一种用于预测药物-靶标相互作用关系的设备的结构示意图。
具体实施方式
下面详细描述本发明的实施例,所述实施例的示例在附图中示出,其中自始至终相同或类似的标号表示相同或类似的元件或具有相同或类似功能的元件。下面通过参考附图描述的实施例是示例性的,仅用于解释本发明,而不能理解为对本发明的限制。
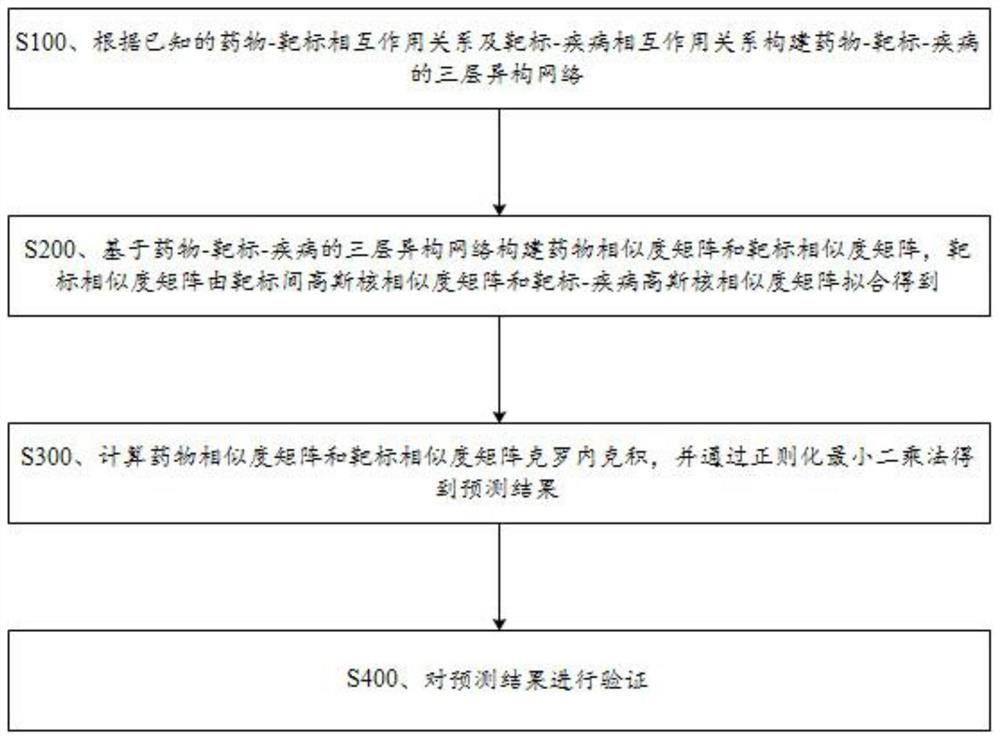
参照图2至图4,本发明的一个实施例,提供了一种用于预测药物-靶标相互作用关系的方法,包括以下步骤:
S100、根据已知的药物-靶标相互作用关系及靶标-疾病相互作用关系构建药物-靶标-疾病的三层异构网络。
其中,药物-靶标相互作用关系可全部由基准数据集提供;也可部分由基准数据集提供,部分由外部数据集(即到目前为止还有机构在补充更新相关信息的外部数据库中提供的数据集)提供;还可全部由外部数据集提供。靶标-疾病相互作用关系由人类疾病关系数据库给出。
首先,需统一所有药物、靶标、疾病的ID,整理成邻接表形式,将邻接表形式的药物-靶标相互作用抽象成药物-靶标的二部图模型;靶标-疾病相互作用也同理抽象成靶标-疾病的二部图模型。在抽象成二部图模型的同时并生成邻接矩阵(用于作为后续正则化最小二乘法的辅助输入),其中有相互作用的矩阵值为1,无相互作用的矩阵值为0。根据药物-靶标的二部图模型和靶标-疾病的二部图模型构建药物-靶标-疾病的三层异构网络。构建三层异构网络的目的是为了维持药物-靶标相互作用和靶标-疾病相互作用维持的整体性,确定药物、靶标和疾病之间的关联关系,以便能够进行后续的拟合和预测处理。如图4,其中Drug={d
S200、基于药物-靶标-疾病的三层异构网络构建药物相似度矩阵和靶标相似度矩阵,靶标相似度矩阵由靶标间高斯核相似度矩阵和靶标-疾病高斯核相似度矩阵拟合得到。
作为一种可选的实施例,药物相似度矩阵由药物化学结构相似度矩阵和药物高斯核相似度矩阵拟合得到。丰富相似度矩阵的类型,能够提升最终的预测结果的准确度。以上四个相似度矩阵的计算过程如下:
第一,药物化学结构相似度矩阵的构建;
首先,计算所有药物的SIMILES(Simplified molecular-input line-entrysystem,简化分子线性输入规范)特征,确保每一种化学结构都有唯一对应的字符串;
然后,将每个SIMILES特征转换为对应的二进制化学指纹;
最后,根据谷本系数方法从所有二进制化学指纹计算得到药物化学结构相似度矩阵。计算公式如下:
其中,f(dx)为药物x的二进制化学指纹。并在此基础上对全部药物构建药物化学结构相似度矩阵。这里二进制化学指纹为166位的二进制位串。
第二,药物高斯核相似度矩阵的构建;
首先,计算所有药物的高斯核参数。高斯核参数定义为空间中任意两点的欧式距离的单调函数;
然后,分别计算每种药物与所有药物之间的高斯核相似度向量,构建所述药物高斯核相似度矩阵。计算公式如下:
K
其中,D={d
第三,靶标间高斯核相似度矩阵的构建;
首先,计算所有靶标间的高斯核参数;
然后,分别计算每个靶标与所有靶标之间的高斯核相似度向量,构建靶标间高斯核相似度矩阵。计算公式如下:
K
其中,T={t
第四,靶标-疾病高斯核相似度矩阵的构建;
首先,计算靶标-疾病的高斯核参数;
然后,分别计算每个靶标与所有疾病之间的高斯核相似度向量,构建靶标-疾病高斯核相似度矩阵。计算公式如下:
K
其中,S={ts
S300、计算药物相似度矩阵和靶标相似度矩阵克罗内克积,并通过正则化最小二乘法得到预测结果。
由于相似度矩阵为非正定方阵并需要将多个相似度矩阵融合到一个大的相似度矩阵中,本步骤S300使用正则化最小二乘法计算矩阵的克罗内克积,具体计算过程分析如下:
首先,将药物化学结构相似度矩阵和药物高斯核相似度矩阵进行线性拟合,将靶标间高斯核相似度矩阵和靶标-疾病高斯核相似度矩阵进行线性拟合,经验化的设置加权因子α=0.5,需要注意的是,α取0.5是较为常用且表示两者具备同等特性的参数比,但不能作为本发明范围的限制。
SIM
SIM
对拟合得到的SIM
W=V∧V
其中,σ为正则化参数,
根据克罗内克积的矩阵方程性质以及克罗内克积转置运算符合分配律,有:
故式(12)可简化为:
令
得到正则化最小二乘法得到预测结果,然后对预测结果进行排序(对矩阵内找最大值及所在行列位置)。
S400、对预测结果进行验证。
作为一种可选的实施方式,步骤S400包括:先用测试集对预测结果进行十倍交叉验证;再引入外部数据集对预测结果进行验证。本验证方法能够有效的体现模型的泛化能力。
仿真实验一:
在上述方法实施的基础上,下表展示了通过本方法得到在酶和GPCR两个数据集上排名最高的(正则化最小二乘法输出值的大小排序)新的药物-靶标相互作用,对预测结果在置信度较高的外部数据库上进行了验证,同时均存在4个药物-靶标相互作用已经被证明存在(下表1和表2中用加粗表示被证明的药物-靶标相互作用):
表1
表2
如表1,在酶的前十名列表中,被验证的靶标来源于Cytochrome P450 2C19。论文《Kirchheiner J,Muller G,Meineke I,Wernecke KD,Roots I,Brockmoller J:Effectsof polymorphisms in CYP2D6,CYP2C9,and CYP2C19 on trimipraminepharmacokinetics.J Clin Psychopharmacol.2003Oct;23(5):459-66.doi:10.1097/01.jcp.0000088909.24613.92》以药物代谢作用等方法进行的实际试验,用化学方法支持了这四个药物-靶标相互作用。表1和表2中未被化学性验证的药物-靶标相互作用具有很高的研究价值。
仿真实验二:
通过多个侧重点不同的数据库来构建三层异构网络模型,并在基准数据集上评估本方法的性能,这些数据集以四种主要的靶标命名,即酶、离子通道、GPCR和核受体,详见论文《Yamanishi Yoshihiro,Araki Michihiro,Gutteridge Alex et al.Prediction ofdrug-target interaction networks from the integration of chemical and genomicspaces.[J].Bioinformatics,2008,24:i232-40》。
表3
其中Drugbank和DisGeNET作为外部数据来源,其他作为验证性数据库使用。
下表4为《Yamanishi Yoshihiro,Araki Michihiro,Gutteridge Alex etal.Prediction of drug-target interaction networks from the integration ofchemical and genomic spaces.[J].Bioinformatics,2008,24:i232-40.》中四个基准数据集中的药物和靶标数量,以及他们的比例。
表4
下表5,使用了《Yamanishi Yoshihiro,Araki Michihiro,Gutteridge Alex etal.Prediction of drug-target interaction networks from the integration ofchemical and genomic spaces.[J].Bioinformatics,2008,24:i232-40.》所提出的同时被广泛使用的外部数据库,并对所得结果在ROC和PR曲线上,对曲线下面积AUC和AUPR进行比较,同时使用的评价标准还包括灵敏度和特异性。由于使用的评价标准较为统一,便不再赘述其来源及计算方法。
表5
其中,FLapRLS方法定义了一个内核,称为高斯交互配置文件(GIP)内核,并使用一个简单的分类器(内核)正则化最小二乘(RLS)来预测药物-靶标相互作用。RLS_Kron使用两种不同的方法首先分别用于构建化学空间和基因组空间,并使用克罗内克积的正则化最小二乘法来预测药物-靶标相互作用。由表5可以看出本方法的预测性能优于FLapRLS方法和RLS_Kron方法。
本方法实施例的有益效果如下:
(1)本方法引入靶标-疾病相互作用关系并结合药物-标靶相互作用关系构建了三层异构网络,基于三层异构网络构建靶标间高斯核相似度矩阵、靶标-疾病高斯核相似度矩阵、药物化学结构相似度矩阵和药物高斯核相似度矩阵,对比传统的预测方法,采用了更为完整的网络结构模型,建立了复杂可靠的相似度矩阵空间,从多个角度预测了全新的药物-靶标相互作用。
(2)本方法使用了克罗内克积的正则化最小二乘法预测最终结果,在计算过程中避免了超规模的矩阵运算。
(3)本方法最终的预测结果通过十倍交叉和外部数据库中得到了较好的验证,通过十倍交叉验证和外部验证法得到本方法的预测性能优于常用的FLapRLS方法和RLS_Kron方法。而且本方法所提出的且未被化学性验证的新的药物-靶标相互作用具有很高的研究价值,其能够使得后续的化学验证试验既可以有针对性的进行,避免大范围没有明确目标的重复性试验。同时因为给出的药物已经通过临床试验等步骤,在最终商业化的重新使用上避免了新研发药物的过长周期。
参照图5,本发明的一个实施例,提供了一种用于预测药物-靶标相互作用关系的设备,该设备包括:一个或多个控制处理器和存储器,图5中以一个控制处理器为例。控制处理器和存储器可以通过总线或者其他方式连接,图5中以通过总线连接为例。
存储器作为一种非暂态计算机可读存储介质,可用于存储非暂态软件程序、非暂态性计算机可执行程序以及模块,如本发明实施例中的设备对应的程序指令/模块。控制处理器通过运行存储在存储器中的非暂态软件程序、指令以及模块,从而实现上述方法实施例所述的用于预测药物-靶标相互作用关系的方法。
存储器可以包括存储程序区和存储数据区,其中,存储程序区可存储操作系统、至少一个功能所需要的应用程序。此外,存储器可以包括高速随机存取存储器,还可以包括非暂态存储器,例如至少一个磁盘存储器件、闪存器件、或其他非暂态固态存储器件。在一些实施方式中,存储器可选包括相对于控制处理器远程设置的存储器,这些远程存储器可以通过网络连接至该用于预测药物-靶标相互作用关系的设备。上述网络的实例包括但不限于互联网、企业内部网、局域网、移动通信网及其组合。
所述一个或者多个模块存储在所述存储器中,当被所述一个或者多个控制处理器执行时,执行上述方法实施例中的用于预测药物-靶标相互作用关系的方法。
本发明实施例还提供了一种计算机可读存储介质,所述计算机可读存储介质存储有计算机可执行指令,该计算机可执行指令被一个或多个控制处理器执行,例如,被图5中的一个控制处理器执行,可使得上述一个或多个控制处理器执行上述方法实施例中的用于预测药物-靶标相互作用关系的方法。
通过以上的实施方式的描述,本领域技术人员可以清楚地了解到各实施方式可借助软件加通用硬件平台的方式来实现。本领域技术人员可以理解实现上述实施例方法中的全部或部分流程是可以通过计算机程序来指令相关的硬件来完成的程序可存储于计算机可读取存储介质中,该程序在执行时,可包括如上述方法的实施例的流程。其中,所述的存储介质可为磁碟、光盘、只读存储记忆体(Read Only Memory,ROM)或随机存储记忆体(Random Access Memory,RAM)等。
在本说明书的描述中,参考术语“一个实施例”、“一些实施例”、“示意性实施例”、“示例”、“具体示例”、或“一些示例”等的描述意指结合该实施例或示例描述的具体特征、结构、材料或者特点包含于本发明的至少一个实施例或示例中。在本说明书中,对上述术语的示意性表述不一定指的是相同的实施例或示例。而且,描述的具体特征、结构、材料或者特点可以在任何的一个或多个实施例或示例中以合适的方式结合。
尽管已经示出和描述了本发明的实施例,本领域的普通技术人员可以理解:在不脱离本发明的原理和宗旨的情况下可以对这些实施例进行多种变化、修改、替换和变型,本发明的范围由权利要求及其等同物限定。
