一种基于多目标优化的协同粒子群SNP相互作用检测方法
文献发布时间:2024-04-18 19:53:33
技术领域
本发明涉及智能计算和数据挖掘领域,基于全基因组关联研究(GWAS)产生的海量单核苷酸多态性(SNP)数据,检测与疾病表型相关的SNP相互作用,对揭示疾病的发病机制和发展趋势具有重要意义。
背景技术
GWAS检测了许多基因组中的数十万个遗传变异,以发现那些与特定性状有统计学关联的变异,并已成为探索复杂疾病遗传结构的常规方法。在GWAS的帮助下,迄今已鉴定出许多与复杂疾病相关的SNP。发现与复杂疾病相关的SNP对揭示疾病的发病机制和发展趋势具有重要意义。然而,它们只能解释复杂疾病中的一小部分潜在遗传机理,并为进一步研究留下了“缺失遗传性”的问题。人们普遍认为,SNP之间的非线性相互作用效应,即SNP相互作用,在解开“缺失遗传力”之谜中起着重要作用。因此,SNP相互作用的检测已经成为GWAS中一个引人注目的步骤。然而,当SNP数量显著增加时,SNP相互作用检测的问题也变得异常复杂。因此,提出了一种有效的基于多目标优化的协同粒子群SNP相互作用检测方法。
针对这类问题,常见的解决算法包括穷举搜索算法和随机搜索算法。穷举搜索算法是在解空间中列出所有可能的SNP组合,然后找到与表型最相关的相互作用。然而,当数据量较大时,即使使用高性能计算的并行硬件架构,完成搜索所需的时间也相当长,从而降低了检测效率。随机搜索算法通过一定的规则从解空间中随机选择候选SNP相互作用,然后评估所选SNP相互作用与表型之间的关联,最终得到与复杂疾病相关的解。与穷举搜索算法相比,随机搜索算法加快了可能与表型相关的SNP相互作用的搜索过程,但存在优化效率低和收敛速度快等缺点。
为了解决以上问题,群智能优化算法因其能够有效地解决优化问题而受到一些学者广泛关注。张等人提出一种基于粒子群的SNP相互作用检测方法,使用无标度网络对方法进行更新,采用互信息作为目标函数。关等人提出一种基于随机分组的自调节人工蜂群方法,采用动态随机分组策略和自调节蜂群优化器检测每个子集的相互作用。上述检测方法均采用单一目标函数来评估SNP相互作用与疾病之间的关联。大量研究表明,这些方法在不同的疾病模型中的检测能力是不稳定的。为了避免单目标检测方法中存在的疾病模型偏好问题,提出了多目标方法来检测相互作用。陈等人提出了一种SNP相互作用检测的多目标遗传方法,以k2分数和基尼指数作为目标函数。顾等人提出了一种基于无标度网络的相互作用检测的多目标人工蜂群方法,通过引入基于对立的学习策略提高了搜索能力。传统的多目标进化方法的缺点是多个目标被同一群体优化,但SNP检测问题受到多个相互冲突的优化目标的约束。因此,本发明提出一种基于多目标优化的协同粒子群SNP相互作用检测方法,基于协同进化框架,利用提出的多种技术,达到检测SNP相互作用的目的。
发明内容
为了克服上述SNP相互作用检测方法的缺陷,本发明提出一种基于多目标优化的协同粒子群SNP相互作用检测方法(CMPSO)。
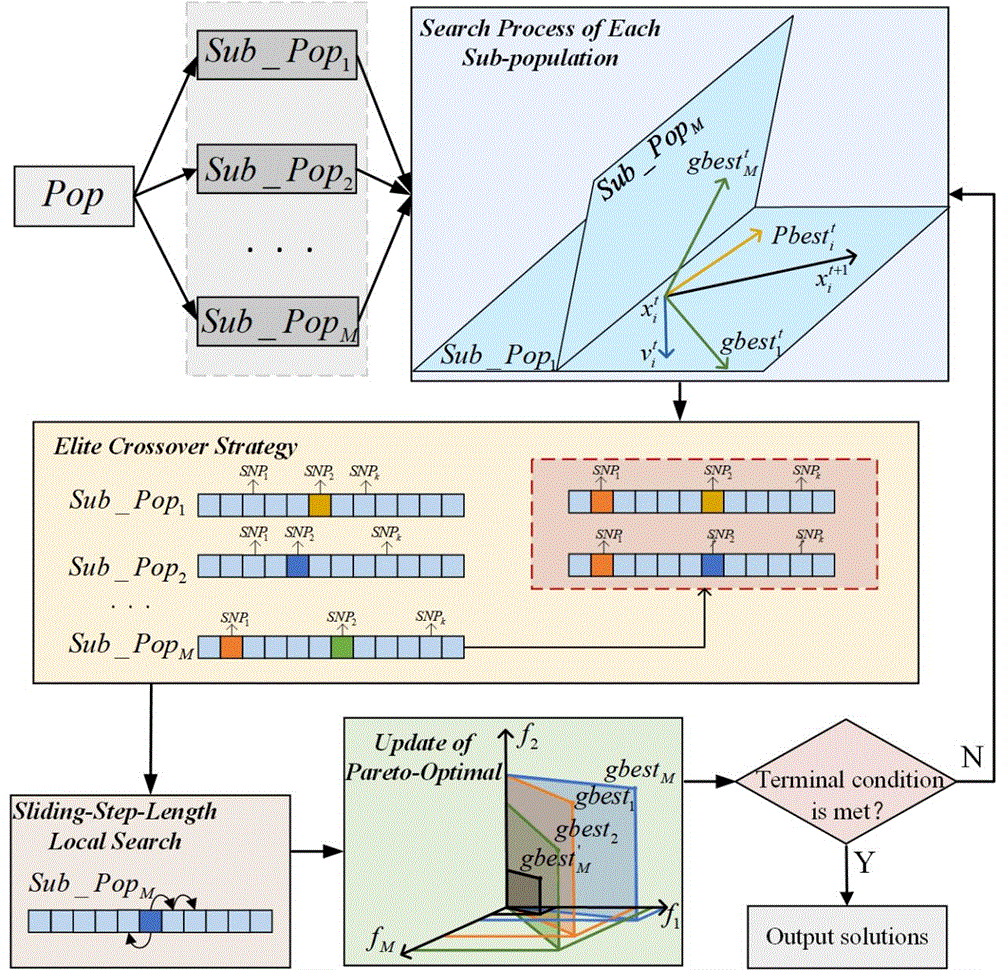
本发明所采用的技术方法是:针对单一目标函数对不同疾病模型具有偏好性的问题,利用协同进化思想划分种群进行SNP相互作用检测方法设计。将原始种群划分为多个子种群,每个子种群使用不同的目标函数独立求解。同时,采用改进的粒子群算法保证了多个子种群之间的信息交互,共同指导粒子的搜索方向。利用基于滑动步长的局部寻优技术降低了方法的迭代次数。最后,通过帕累托最优策略与划分种群技术的结合,有效降低了解的假阳性,得到与疾病表型最相关的SNP相互作用。
附图说明
下面将结合附图及实施实例对本发明作进一步的说明,附图中:
图1是本发明方法检测SNP相互作用的流程示意图。
图2是划分种群独立求解技术的具体实施过程伪代码。
图3是基于滑动步长的局部寻优技术的具体实施过程的伪代码。
图4是仿真数据使用的模型外显率表格。每个模型分别生成100、1000、10000三个不同维度SNP的数据,每个模型包含100个数据集,每个数据集包含2000个致病样本和2000个对照样本。
图5-7是本发明在仿真数据集上的精度实验结果示意图。仿真数据是基于不同模型使用仿真软件生成的100、1000、10000维的SNP数据。本发明与其他7种同类方法的精确度比较。每种方法在每个模型上独立运行50次,取其平均值。
图8-10是本发明在仿真数据集上的时间实验结果示意图。
图11是本发明在年龄相关性黄斑变性真实疾病数据集上的实验结果示意图。
具体实施方式
为了使本发明的目的、技术方案及优点更加清晰明了,结合以下步骤和图1对本发明进行进一步的详细描述。需要说明的是,本实例以本技术方案为前提,给出详细的实施步骤和具体的操作方式,但本发明并不限于本实例。
参照图1,本发明的方法简称为CMPSO,其具体的实现步骤如下:
步骤1,对SNP数据进行预处理:
(1.1)真实数据:对每个SNP位点进行编码,当对应位点上的两个等位基因为纯合子AA时取l,纯合子aa时取2,杂合子Aa或aA时取3,当对应位点上的等位基因数据缺失时取0。为了减少丢失数据带来的信息损失,采用了最近邻方法处理年龄相关性黄斑变性真实数据的丢失信息。样本类标0表示对照组,1表示疾病组,数据样本总数记为N,数据维度记为D。
(1.2)仿真数据:计算疾病模型,通过仿真软件EpiSIM分别生成包含100、1000、10000个SNP的数据。对于每个数据集,只有一个SNP相互作用与表型相关,其它SNP与表型无关。生成的数据矩阵,行为样本,列为SNP,编码方式与真实数据相同。
步骤2,初始化原始种群及变量:
(2.1)设置CMPSO方法相关参数,包括种群规模
(2.2)在解空间中根据原始的粒子群算法随机初始化原始种群
(2.3)设置粒子群算法相关参数
步骤3,原始种群划分:
(3.1)设置循环,从1到
(3.2)将原始种群随机划分为
(3.3)每个子种群对应于一个目标函数独立求解;
(3.4)根据子种群对应的目标函数计算种群中粒子的适应度值(每个粒子对应一个SNP相互作用),每个种群中适应度值最大的粒子称为领导者,其余粒子为学习者,将所有领导者存放在外部学习库
(3.5)每个子种群的粒子根据改进的粒子群算法更新。
步骤4,子种群搜索和更新
(4.1)每个子种群中的所有粒子通过以下学习公式向所有子种群的领导者学习,
(4.2)更新粒子速度之后根据以下公式更新每个粒子的位置
(4.3)计算每个种群中所有粒子的适应度值,将每个子种群的最大适应度值与对应外部学习库的领导者的适应度值比较,若大于,则更新外部学习库的领导者;
(4.4)将每个子种群的领导者进行交叉操作,即把领导者位置不同位点进行笛卡尔积运算,得到新的个体,若新的解优于原来的领导者则更新。
步骤5,基于滑动步长的局部寻优技术
(5.1)所有子种群粒子学习更新后,借助基于滑动步长的局部寻优技术获得全局最优解;
(5.2)比较每个子种群的领导者是否找到了相同的位点;
(5.3)所有的领导者之间的距离用
(5.4)以range为步长,向前和向后滑动进行局部搜索。
步骤6,帕累托最优策略
(6.1)每个种群得到适应度值最大的解须满足帕累托最优策略才进行更新,有效解决了真实数据对于某个目标函数偏好性的问题,降低了解的假阳性;
(6.2)
重复步骤3、4、5、6,直到迭代结束,得到最优的SNP相互作用组合,即为全局最优解。
本发明将通过下述的实例对本发明方法的效果进行更详细的描述,这些实例仅用于举例目的,而不试图限制本发明的应用范围。
实验1:仿真数据中SNP相互作用的识别
通过疾病模型生成100、1000、10000维度不同的24组数据记为100-SNP数据、1000-SNP数据、10000-SNP数据。每个数据集包含2000个病例样本,2000个对照样本,只有一组SNP相互作用与疾病表型相关,其余无关。
表1是8个疾病模型的外显率表格。
表1
对生成的仿真数据与7种典型的SNP相互作用检测方法(SIPSO、MACOED、RCABC、SFMOABC、PSOMiner、IOBLPSO、MACOED)进行比较,采用精度与运行时间两个评价指标,精度实验结果于说明书附图中图5-图7展示,运行时间实验结果于说明书附图中图8-图10展示。由实验结果图可以看出,本发明方法实验结果比较稳定,有较高的检测精度,与其他方法精度相当的情况下,有较快的运行速度,表明了方法的有效性。
实验2:在真实数据上的SNP相互作用检测
真实数据使用年龄相关性黄斑变性(AMD)数据。AMD数据包含146个样本,其中96个病例样本,50个对照样本,包含103611个SNP。样本数值为0、1、2、3,其中0表示数据丢失,为了减少丢失数据带来的信息损失,采用最近邻方法处理AMD数据丢失信息。
表2展示的是使用CMPSO方法找到的SNP相互作用
表2
其中,多数SNP相互作用与其他方法找到的SNP相互作用相同。例如:(rs380390,rs1363688)被CINOEDV、EpiACO、SFMOABC、IOBLPSO、MP-HS-DHSI等方法验证。CMPSO还给出了其他方法没有找出但是本发明找到的与疾病表型关联度很高的SNP相互作用,不排除它们与疾病真正关联的可能性。
