生物分子结构弛豫可视化交互方法及装置
文献发布时间:2024-01-17 01:27:33
技术领域
本发明涉及生物分子信息技术领域,尤其涉及生物分子结构弛豫可视化交互方法及装置。
背景技术
生物分子结构决定生物分子的性质和功能,对于生物分子的稳定天然态结构,实验上可以借助X射线晶体衍射和冷冻电镜等技术给出高分辨率原子位置信息。由于X射线晶体衍射和冷冻电镜无法直接提供分子运动的动态信息,因此,对分子运动的实时动态信息的研究非常匮乏,使得很多对生物分子的更多性能研究停留在最初设计的固定的分子结构上,未能深入研究潜在的分子弛豫过程及弛豫之后分子结构的改变,由此极大限制了药物设计和发现能力,以及降低了药物设计的精确性。
发明内容
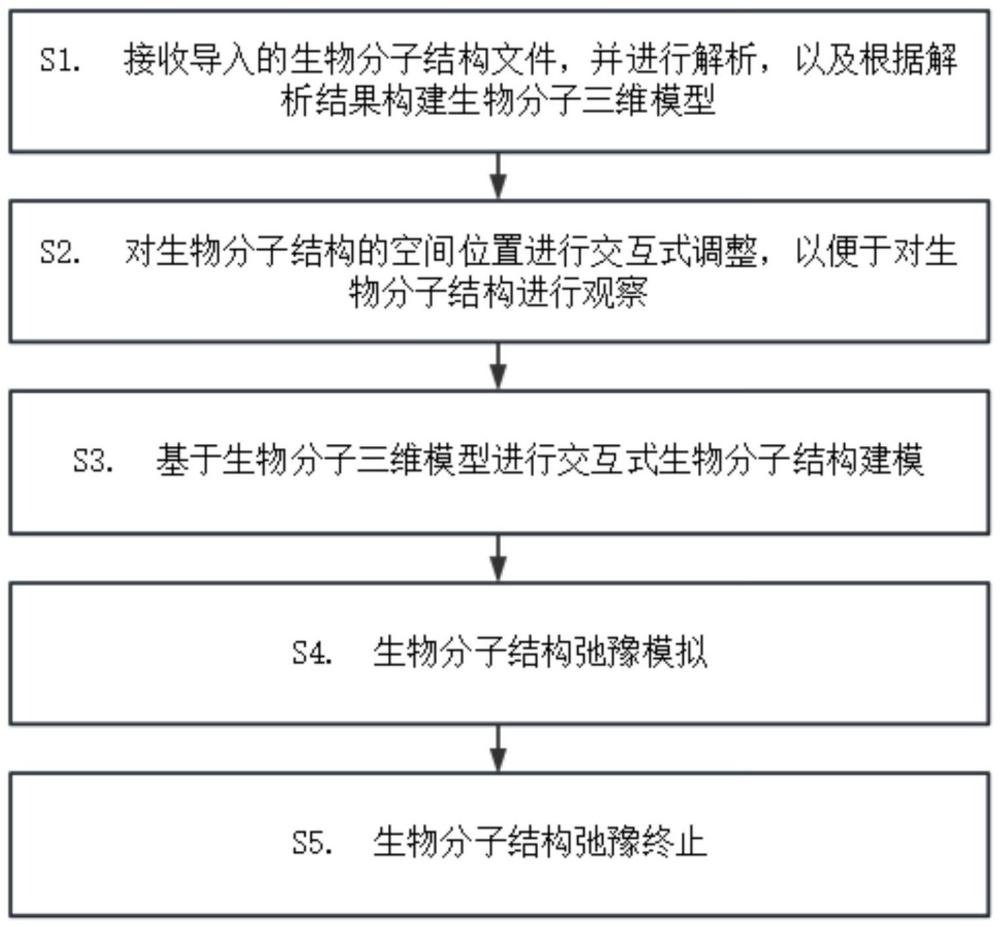
针对现有技术,本申请提出生物分子结构弛豫可视化交互方法,其包括:
S1.接收导入的生物分子结构文件,并进行解析,以及根据解析结果构建生物分子三维模型;
S2.对生物分子结构的空间位置进行交互式调整,以便于对生物分子结构进行观察;
S3.基于生物分子三维模型进行交互式生物分子结构建模;
S4.生物分子结构弛豫模拟;
S5.生物分子结构弛豫终止。
根据本发明一些实施例,所述的方法还包括基于以下设计来实施步骤S4:
力学模型:力学环境包括共价键连接原子间的键长伸缩势能和非共价键连接原子间的兰纳-琼斯(Lennard-Jones)势能;共价键伸缩势能采用理想弹簧模型,通过虎克定律(Hooke’s law)将共价键的拉升变形与原子受力定量为线性关系;
加速度计算:F(t)=ma(t),其中,F(t)为t时刻原子所受净力,m为原子质量,a(t)为t时刻原子加速度;
时间演化算法:引入Verlet算法进行各原子位置坐标和速度的迭代计算和更新;Verlet算法的基本原理是根据牛顿定律计算原子的位置和速度,采用离散时间补偿进行时间演化;
位置更新:r(t+Δt)=2r(t)-r(t-Δt)+a(t)×t
速度更新:v(t)=[r(t+Δt)-r(t-Δt)]/2Δt。
根据本发明一些实施例,所述的方法还包括:以文件形式保存生物分子结构弛豫的动力学轨迹。
根据本发明一些实施例,所述的方法还包括:对结构弛豫轨迹的间隔时间进行设定,根据间隔时间,生成生物分子结构弛豫的动力学轨迹文件中的数据。
根据本发明一些实施例,对生物分子结构的空间位置进行交互式调整,包括,控制观察视角的拉近、推远、平移和旋转,以实现生物分子结构的放大和缩小以及视角切换。
根据本发明一些实施例,所述基于生物分子三维模型进行交互式生物分子结构建模包括通过实施单原子或多原子的抓取、平移和旋转操作,以改变生物分子结构。
根据本发明一些实施例,生物分子结构弛豫终止包括当确定某个时间段内的结构调整幅度小于设定阈值时,选择终止结构弛豫过程,或者在某个时间段内的结构调整幅度小于设定阈值时,自动终止结构弛豫过程。
根据本发明一些实施例,所述的方法还包括:基于虚拟现实实施所述方法。
本申请还提出一种生物分子结构弛豫可视化与交互装置,其包括:计算设备、交互设备、显示设备和传感器设备;计算设备用于执行所述的方法;显示设备用于呈现交互画面;交互设备用于实施交互操作;传感器设备用于监测交互设备的交互动作。
本申请还提出另一种生物分子结构弛豫可视化交互装置,其所述的生物分子结构弛豫可视化交互装置,包括:计算设备、头戴式虚拟现实设备、虚拟现实手柄设备和传感器设备;计算设备用于执行所述的方法;头戴式虚拟现实设备用于呈现交互画面;虚拟现实手柄设备用于实施交互操作;传感器设备用于监测虚拟现实手柄设备的交互动作。
生物分子结构是复杂力学的综合结果,结构不同,相互作用就有差异。本发明的交互式结构建模通过有目的的生物分子结构设计和相互作用识别来挖掘和建立生物分子定量构效关系,以及还进一步考虑到交互式结构建模可能会引入的不合理结构,通过构建力学模型对交互式结构建模生成的新结构在力学作用下进行的结构微调整和折叠过程(弛豫过程)进行了研究,使得对生物分子的更多性能研究不再停留在最初设计的分子结构上,而是深入到分子结构微调和生物分子形状改变后的分子结构,从而对生物分子结构数据库进行了有效扩展,为基于生物大数据的人工智能应用节省了大量的人力、物力和财力,并极大地提高了智能计算性能,提升药物设计和发现能力。
附图说明
图1为根据本发明一些实施例的生物分子结构弛豫可视化交互装置的示意图。
附图标记说明:21-计算设备;20右手柄;22-左手柄;23-传感器设备;25-头戴式虚拟现实设备。
图2a和图2b分别为根据本发明一些实施例的左手柄和右手柄的结构示意图。
附图标记说明:1-菜单按钮;2-触控板;3-系统按钮;4-状态指示灯;5-Micro-USB端口;6-扳机;7-手柄按钮。
图3a为根据本发明的一些实施例的生物分子结构弛豫可视化交互方法流程图。
图3b为根据本发明的一些实施例的图3a的步骤S1的实现流程图。
图4a和图4b分别为根据本发明的一些实施例的虚拟现实生物分子三维建模过程示意图。
图5a,5b,5c,5d为根据本发明的一些实施例的对虚拟现实场景内的生物分子结构的空间位置进行沉浸式调整的示意图。
图6为根据本发明的一些实施例的虚拟现实交互式生物分子结构建模示意图。
图7为根据本发明的一些实施例的虚拟现实生物分子结构弛豫模拟示意图。
具体实施方式
下面将结合附图和本发明实施例,对依据本发明的具体实施方案、结构及特征进行清楚、完整地描述。
图1为根据本发明一些实施例的生物分子结构弛豫可视化交互装置的示意图。
所述装置主要包括计算设备21、头戴式虚拟现实设备25、虚拟现实手柄设备(或简称手柄设备,图中示出左手柄22和右手柄20,根据实际需要,也可以将两个手柄设备的功能集成到一个手柄设备上)和传感器设备23(也可以配置左右两个传感器设备)等。计算设备为主控制器,用于执行以下步骤S1、S2、S3、S4和S5及其他步骤。头戴式虚拟现实设备用于呈现交互画面,如虚拟现实场景中的生物分子三维模型,及其他交互过程中出现的画面。虚拟现实手柄设备用于实施交互操作。传感器设备用于监测交互动作,例如,监测手柄设备的位置信号和按键操作。
图2a和图2b分别为根据本发明一些实施例的左手柄和右手柄的结构示意图。
附图标记说明:1-菜单按钮;2-触控板;3-系统按钮;4-状态指示灯;5-Micro-USB端口;6-扳机;7-手柄按钮。
左右手柄上的触控板2、扳机6和系统按钮3等部件与虚拟现实场景中的原子(团)抓取、观察视角拉近和推远以及平移和旋转等交互功能匹配。使用者通过左右手柄操作触发相应功能,实现对生物分子在虚拟现实场景的交互功能。状态指示灯4可以用于指示当前的分子状态,例如弛豫阶段或弛豫终止阶段等。Micro-USB端口5可以用于和计算设备21或其他pc连接,以便计算设备21或其他pc对手柄的各个按钮的对应功能进行设置或调整。
图3a为根据本发明的一些实施例的生物分子结构弛豫可视化交互方法流程图,所述方法的各步骤可以由所述计算设备21来执行。如图所示,所述方法包括以下步骤S1、S2、S3、S4和S5:
步骤S1:接收导入的生物分子结构文件,并进行解析,以及根据解析结果构建生物分子三维模型。
生物分子结构文件可以包括PDB和mol等格式,根据本发明的一些实施例,如图3b所示,步骤S1可以包括:
S11.接收PDB文件;
S12.解析PDB文件;
S13.根据解析结果,构建原子球体模型;
S14.根据解析结果,构建共价键棒体模型。
S15.完成三维建模。
在以上步骤中,原子用球体模型表征。球体的大小和颜色可由原子类型设定。分子内化学共价键的识别可以由Open Babel开源化学信息学软件完成。共价键可以用棒体模型表征。棒体的大小和颜色可由共价键类型设定。
考虑到生物分子所包含原子数差异,在本发明的一些实施例中,在进行生物分子三维建模过程中,可以使球体和棒体的初始尺寸随虚拟现实场景布局进行等比例调整,以优化视觉效果。
图4a和图4b为根据本发明的一些实施例的虚拟现实生物分子三维建模过程示意图。
在本实施例中,如图4a所示,加载的生物分子由7个原子组成,其中,红色球体为氧原子,黄色球体为氮原子,黑色球体为碳原子。识别的共价键共有7个,采用默认的灰色棒体标注,如图4b所示。
步骤S2:对生物分子结构的空间位置进行交互式调整,以便于对生物分子结构进行观察;
在该步骤中,使用者可以通过右手柄的触控板控制虚拟现实观察视角的拉近、推远、平移和旋转功能,实现生物分子结构的放大和缩小以及视角切换,方便使用者进行沉浸式生物分子结构调整。
放大和缩小:放大和缩小功能是通过右手柄上的触控板2和左手柄上扳机6共同操作完成。使用者可以在按下左手柄扳机6的同时,通过右手柄20触控板2的“上”和“下”触控控制虚拟现实观察视角的“前进”和“后退”,拉近和推远生物分子,以及实现生物分子结构的放大和缩小等功能。
观察视角的平移:观察视角的平移功能是通过右手柄触控板2操作完成。使用者通过右手柄触控板的“上”、“下”、“左”和“右”按键控制观察视角的四个方向移动,完成观察视角的平移操作。
观察视角的旋转:观察视角的选择功能是通过右手柄触控板2操作完成。使用者通过右手柄触控板的“上”、“下”、“左”和“右”按键控制观察视角的四个方向移动,完成观察视角的旋转操作。
图5a,5b,5c,5d为根据本发明的一些实施例的对虚拟现实场景内的生物分子结构的空间位置进行沉浸式调整的示意图。
在本实施例中,通过手柄设备的触控板2和扳机6按键控制观察视角的移动,以便于对生物分子结构进行观察,例如,实现对生物分子结构的放大和缩小以及多角度观察功能。
步骤S3:基于生物分子三维模型进行交互式生物分子结构建模。
根据本发明的一些实施例,在该步骤中,使用者可以通过左手柄扳机6实现单原子或多原子的抓取、平移和旋转等功能,改变生物分子结构。
原子(团)抓取:原子(团)抓取功能的实现是通过扳机6操作完成。使用者移动右手柄至拟抓取的原子附近,按下扳机6实现对该原子的抓取操作。如需对多个原子进行抓取,使用者在按下左手柄扳机6的同时按下右手柄系统按钮3,可将该原子添加到已抓起的原子团中。
原子(团)平移:原子(团)平移功能的实现是通过感应右手柄移动轨迹完成。在使用者通过右手柄完成对原子(团)的抓取操作后,移动右手柄,计算设备根据传感器监测到的右手柄移动轨迹实现原子(团)的平移操作。
原子(团)旋转:原子(团)旋转功能的实现是通过感应右手柄旋转轨迹完成。在使用者通过右手柄完成对原子(团)的抓取操作后,按下左手柄的手柄按钮7的同时旋转右手柄,计算设备根据传感器监测到的右手柄旋转轨迹实现原子(团)的旋转操作。
步骤S2和S3分别对应结构观察调整模式和结构建模模式。这两种模式下手柄均有平移和旋转操作。在结构观察模式下,平移和旋转操作仅用于切换视觉效果,不改变生物分子结构。在结构建模模式下,平移和旋转操作是对所抓取的原子(团)的位置调整,改变生物分子结构。使用者可根据需要通过左右手柄上的按键进行交互模式选择。
以上对各个按键功能的描述和按键组合方式还可以采用其他各种方式。
图6为根据本发明的一些实施例的交互式生物分子结构建模示意图。
在本实施例中,通过左手柄扳机6对所加载生物分子中的两个原子进行抓取和平移操作。根据本发明的一些实施例,所述共价键模式可以包括可断开模式和不可断开模式。图6分别给出了两种模式的共价键监听结果。
在不可断开模式下,这些被识别的共价键不会断开,也不会有新的共价键形成。在可断开模式下,实时监听原子间距离信息,实现共价键形成和断开的动态调整。使用者可根据需要通过手柄设备进行共价键模式选择。
步骤S4:生物分子结构弛豫模拟。
发明人注意到,交互式结构建模会引入不合理结构,生物分子在力学作用下进行持续的结构微调整,折叠形成稳定的新结构,以消除初始的不合理结构。
在本发明中,使用者在完成交互式结构建模后,通过左手柄的一个手柄按钮7触发结构弛豫功能。在结构弛豫过程中,共价键处于不可断开模式。使用者在使用头戴式虚拟现实设备观察的同时,也可以操作手柄进行观察视角的切换。
根据本发明的一些实施例,基于以下设计来实施步骤S4:
力学模型:力学环境包括共价键连接原子间的键长伸缩势能和非共价键连接原子间的兰纳-琼斯(Lennard-Jones)势能;共价键伸缩势能采用理想弹簧模型,通过虎克定律(Hooke’s law)将共价键的拉升变形与原子受力定量为线性关系;兰纳-琼斯势能以两原子间距离为唯一变量,结合近距离时的互相排斥作用和远距离的相互吸引作用。
加速度计算:F(t)=ma(t),其中,F(t)为t时刻原子所受净力,m为原子质量,a(t)为t时刻原子加速度。
时间演化算法:引入Verlet算法进行各原子位置坐标和速度的迭代计算和更新;Verlet算法的基本原理是根据牛顿定律计算原子的位置和速度,采用离散时间补偿进行时间演化。
位置更新:r(t+Δt)=2r(t)-r(t-Δt)+a(t)Δt
速度更新:v(t)=[r(t+Δt)-r(t-Δt)]/2Δt。
图7为根据本发明的一些实施例的生物分子结构弛豫示意图。
如图7所示,结构建模完成后的生物分子在力学作用下,出现结构弛豫,进行一系列的结构微调整,以消除交互建模中的不合理结构,最终折叠形成稳定的新结构。
步骤S5:生物分子结构弛豫终止;
在该步骤中,使用者可通过右手柄的菜单按钮1终止结构弛豫。根据本发明的一些实施例,当计算设备确定在某个时间段内的结构调整幅度小于设定阈值时,可选择终止结构弛豫,或者可以进行自动设定,使得在某个时间段内的结构调整幅度小于设定阈值时,自动终止结构弛豫过程。
根据本发明的一些实施例,所述方法还可以进一步包括步骤:以文件形式保存生物分子结构弛豫的动力学轨迹,用于后续分析。根据本发明的一些实施例,使用者还可通过手柄操作对结构弛豫轨迹的间隔时间进行设定,例如,以t为单位,生成生物分子结构弛豫的动力学轨迹文件中的数据。
以上描述了基于虚拟现实的生物分子结构弛豫可视化交互装置及生物分子结构弛豫可视化交互方法,根据实际需要,所述交互还可以是基于pc和显示器的显示交互技术。相应地,生物分子结构弛豫可视化交互装置可以包括:计算设备、交互设备、显示设备和传感器设备;计算设备用于执行所述的方法步骤;显示设备用于呈现交互画面;交互设备用于实施交互操作;传感器设备用于监测交互设备的交互动作。
生物分子结构是复杂力学的综合结果,结构不同,相互作用就有差异。本发明的交互式结构建模通过有目的的生物分子结构设计和相互作用识别来挖掘和建立生物分子定量构效关系,以及还进一步考虑到交互式结构建模可能会引入的不合理结构,通过构建力学模型对交互式结构建模生成的新结构在力学作用下进行的结构微调整和折叠过程(弛豫过程)进行了研究,使得对生物分子的更多性能研究不再停留在最初设计的分子结构上,而是深入到分子结构微调和生物分子形状改变后的分子结构,从而对生物分子结构数据库进行了有效扩展,为基于生物大数据的人工智能应用节省了大量的人力、物力和财力,并极大地提高了智能计算性能,提升药物设计和发现能力。
以上所述,仅是本发明的较佳实施例而已,并非对本发明作任何形式上的限制。基于本发明中的实施例,任何本领域技术人员在没有作出创造性劳动前提下所获得的所有其他实施例,都属于本发明保护的范围。
- 生物信息可视化装置及方法
- 基于JavaScript的交互式网页生物大数据可视化方法
- 面向可视化视图构建的徒手交互方法、可视化视图构建装置和存储介质
