一种TB级生物医学三维图像数据的大数据格式归档方法
文献发布时间:2023-06-19 19:30:30
技术领域
本发明属于生物医学领域,更具体地,涉及一种TB级生物医学三维图像数据的大数据格式归档方法。
背景技术
TB级生物医学三维图像数据大数据格式的制作,是对原始图像序列,进行不同分辨率切片,大数据格式0级为最高分辨率,三维块数量最多,每递增1级,由上一级x、y、z三个方向的两两三维块合并,然后三个方向各下采样2倍,生成一个新的三维块,分辨率降低8倍,三维块数量减少8倍。三维块便于后续可视化时进行快速虚拟坐标块访问,多分辨率用于可视化交互时大视野用低分辨率图像,小视野用高分辨率图像,从而满足实时三维交互的可视化需求。
其中,在进行切片时是逐帧读取图像序列,对每一帧进行二维切片,然后压缩图像写入大数据格式0级分辨率对应的三维块中,再对这帧图像进行下采样、二维切片、压缩后写入大数据格式1级对应的三维块中,相同的方法再制作剩余的2级、3级、……、n级大数据格式,大数据格式最大级别n可人为设置,也可根据需求计算得到,如到某一级别x、y、z轴方向最小像素长度小于某一值时,为最大大数据格式级别n;对图像序列中的其它帧,依次进行上述处理后,得到制作好的大数据格式。
但现有制作方法压缩时间较长,压缩比较低,压缩后的解压缩速度也较慢,解压速度慢导致后续可视化只能查看较低分辨率图像,查看高分辨率图像时,需等待几秒钟,用户体验感较差;其次大数据图像序列制作时,图像尺寸较大,全部读进内存,占用设备计算内存较多,对整张图切片再保存到对应的三维小块中,会导致打开的三维小块文件过多,IO稳定性差,容易崩溃,每一个小块存储地址都不连续,导致大部分写入都是随机写入,性能较慢;其次每读一张图片需要几秒钟的时间,这段时间仅用来读图,其他资源利用率不高,制作效率较低。现有方案一般采用集群来解决TB级生物医学三维图像数据大数据格式制作时间久、效率低、容易崩溃等问题,但集群价格昂贵,搭建复杂,难以普遍应用。
发明内容
针对现有技术的缺陷和改进需求,本发明提供了一种TB级生物医学三维图像数据的大数据格式归档方法,其目的在于提供一种成本低且解压缩速度快的TB级生物医学三维图像数据的大数据格式归档方法。
为实现上述目的,按照本发明的一个方面,提供了一种TB级生物医学三维图像数据的大数据格式归档方法,包括:
对TB级生物医学二维图像序列中各帧二维图像进行切片,其中,各帧二维图像的切片信息一致,且所述切片信息包括:切片数量、切片尺寸和各切片位置坐标;
对每帧二维图像进行区域划分以将切片分组,各帧二维图像的区域划分数量、尺寸和位置坐标一致;将各帧二维图像之间每个相同位置坐标对应的区域叠加,得到每个位置坐标对应的三维局部块,该三维局部块的长度为所述TB级生物医学二维图像序列的长度,宽度和高度为对应区域的宽度和长度;
分别独立地读取各三维局部块至内存,以分别对各三维局部块进行切块,得到若干个三维小块,每个三维小块的长度为所述TB级生物医学二维图像序列的长度,宽度和高度为一个或多个切片的高度和宽度;利用每个三维小块z轴方向连续变化的信息,对该三维小块进行压缩,将压缩数据写入该三维小块对应的存储容器中,完成对TB级生物医学二维图像序列的0级大数据格式归档,其中,所述z轴方向表示TB级生物医学二维图像序列排列方向。
进一步,还包括:对每个三维局部块进行n次下采样,分别独立地读取下采样后的各三维局部块,以分别对下采样后的各三维局部块进行切块,得到若干个三维小块,每个三维小块的长度为所述TB级生物医学二维图像序列的长度,宽度和高度为一个或多个切片的高度和宽度;利用每个三维小块z轴方向连续变化的信息,对该三维小块进行压缩,将压缩数据写入该三维小块对应的存储容器中,完成对所述TB级生物医学二维图像序列的n级大数据格式归档,n为正整数。
进一步,在对TB级生物医学二维图像序列中各帧二维图像进行切片之前,还包括:计算TB级生物医学二维图像序列中每帧二维图像的0级大数据格式所对应的切片信息以及三维局部块尺寸,具体为:
(1)计算每帧二维图像在x、y方向切片数量nx0和ny0,计算方式为:
其中,
(2)假设每一切片的x、y方向序号分别为idx0、idy0,idx0为x方向第idx0+1块,idy0为y方向第idy0+1块,则当前切片起点坐标为startPx0、startPy0,终点坐标为endPx0、endPy0,计算公式为:
其中min()为最小值;
(3)三维局部块尺寸计算方式如下:
其中,sw和sh分别为三维局部块高和宽。
进一步,还包括:计算TB级生物医学二维图像序列中每帧二维图像的n级大数据格式所对应的切片信息以及三维局部块尺寸,具体为:
(1)计算1级大数据格式对应的三维小块高和宽分别为h1、w1,计算方式为:
(2)计算1级大数据格式对应的每帧二维图像在x、y方向切片数量nx0和ny0,计算方式为:
其中,
(3)假设每一切片的x、y方向序号分别为idx1、idy1,idx1为x方向第idx1+1块,idy1为y方向第idy1+1块,则当前切片起点坐标为startPx1、startPy1,终点坐标为endPx1、endPy1,计算公式如下:
其中,min()为最小值。
(4)其它级大数据格式同1级大数据格式没有冗余,以相同的参数计算方式计算对应的切片信息以及三维局部块尺寸。
进一步,所述利用每个三维小块z轴方向连续变化的信息,对该三维小块进行压缩,具体实现方式为:
通过三维小块图像数据的z轴方向连续性特征,使用三维小块中第一帧加上第二帧与第一帧的变化信息表示第二帧,第二帧加上第三帧与第二帧的变化信息表示第三帧,以此类推,实现对三维小块的压缩。
进一步,在对当前级大数据格式对应的各三维小块进行逐帧压缩的同时,每集齐三维局部块中连续两帧二维图像,即进行最大值采样,最终生成下一级大数据格式对应的三维局部块。
进一步,在分别独立地读取各三维局部块至内存时,每个三维局部块的读取方式为:依次串行读取三维局部块的每一帧,同时并行解码所读取的每一帧数据。
本发明还提供一种计算机可读存储介质,所述计算机可读存储介质包括存储的计算机程序,其中,在所述计算机程序被处理器运行时控制所述存储介质所在设备执行如上所述的一种TB级生物医学三维图像数据的大数据格式归档方法。
总体而言,通过本发明所构思的以上技术方案,能够取得以下有益效果:
(1)本发明方法将生物医学三维图像z轴的连续性,用于三维图像的压缩,同时提出直接快速读取局部图像方法,实现TB级生物医学三维图像数据大数据格式的快速制作。具体地,计算大数据图像的切块信息;直接快速读取局部块,无论三维图像序列尺寸如何,都可把原始图像划分为一个个完全独立处理的局部块,使其适用于所有尺寸的TB级生物医学三维图像数据大数据格式的制作,其次局部块尺寸不大,相对于直接处理原始图像,IO稳定性强,连续写入操作多,写入速度快;在内存中结合计算的切块信息对局部块进行切片,最后利用z轴连续性特征对图像进行压缩并连续写入对应的大数据格式的存储容器中,实现TB级生物医学三维图像数据大数据格式的快速制作。该方法制作速度快,压缩比高,并且解压缩即还原图像速度也很快,稳定性强,可解决现有技术制作存在的高成本、易用性差、实用性低等问题。
(2)读取三维局部块时,相对于传统方法一帧分为多个部分,每次读取一部分,然后解码,再读另一部分,当并行读图时,会有大量的随机读取,对于硬盘随机读取性能很低,读图速度慢。本发明采用依次串行读取三维局部块的每一帧,同时并行解码读取的每一帧数据的方法,极大的降低了读图时随机读取的次数,理论上达到硬盘读取速度极限。
(3)本发明方法有较高的并行处理可行性。在每级大数据格式制作中,各三维局部块可并行进行三维小块切块和数据压缩,另外,在由三维局部块切块至三维小块的过程中以及由三维小块到数据压缩的过程中,都可以并行进行下采样以进行下一级大数据格式的制作,极大提高了归档速度。
附图说明
图1为本发明实施例提供的一种TB级生物医学三维图像数据的大数据格式归档方法流程框图;
图2为本发明实施例提供的一种TB级生物医学三维图像数据的大数据格式归档方法流程示意图。
具体实施方式
为了使本发明的目的、技术方案及优点更加清楚明白,以下结合附图及实施例,对本发明进行进一步详细说明。应当理解,此处所描述的具体实施例仅仅用以解释本发明,并不用于限定本发明。此外,下面所描述的本发明各个实施方式中所涉及到的技术特征只要彼此之间未构成冲突就可以相互组合。
实施例一
一种TB级生物医学三维图像数据的大数据格式归档方法,如图1所示,包括:
对TB级生物医学二维图像序列中各帧二维图像进行切片,其中,各帧二维图像的切片信息一致,且切片信息包括:切片数量、切片尺寸和各切片位置坐标;
对每帧二维图像进行区域划分以将切片分组,各帧二维图像的区域划分数量、尺寸和位置坐标一致,各帧之间相同位置坐标对应区域的切片数量、尺寸和位置坐标相同;将各帧二维图像之间每个相同位置坐标对应的区域叠加,得到每个位置坐标对应的三维局部块,该三维局部块的长度为所述TB级生物医学二维图像序列的长度,宽度和高度为对应区域的宽度和长度;
分别独立地读取各三维局部块至内存,以分别对各三维局部块进行切块,得到若干个三维小块,每个三维小块的长度为TB级生物医学二维图像序列的长度,宽度和高度为一个或多个切片的高度和宽度;利用每个三维小块z轴方向连续变化的信息,对该三维小块进行压缩,将压缩数据写入该三维小块对应的存储容器中,完成对TB级生物医学二维图像序列的0级大数据格式归档,其中,z轴方向表示TB级生物医学二维图像序列排列方向。
可作为优选的实施方案,方法还包括:
对每个三维局部块进行n次下采样,分别独立地读取下采样后的各三维局部块,以分别对下采样后的各三维局部块进行切块,得到若干个三维小块,每个三维小块的长度为所述TB级生物医学二维图像序列的长度,宽度和高度为一个或多个切片的高度和宽度;利用每个三维小块z轴方向连续变化的信息,对该三维小块进行压缩,将压缩数据写入该三维小块对应的存储容器中,完成对所述TB级生物医学二维图像序列的n级大数据格式归档,n为正整数。
需要说明的是,在对TB级生物医学二维图像序列中各帧二维图像进行切片之前,还包括:计算TB级生物医学二维图像序列中每帧二维图像的0级大数据格式所对应的切片信息以及三维局部块尺寸,可作为优选的实施方案,计算方式具体为:
(1)计算每帧二维图像在x、y方向切片数量nx0和ny0,计算方式为:
其中,
(2)假设每一切片的x、y方向序号分别为idx0、idy0,idx0为x方向第idx0+1块,idy0为y方向第idy0+1块,则当前切片起点坐标为startPx0、startPy0,终点坐标为endPx0、endPy0,计算公式为:
其中min()为最小值;
(3)三维局部块尺寸计算方式如下:
其中,sw和sh分别为三维局部块高和宽。
另外,方法还包括:计算TB级生物医学二维图像序列中每帧二维图像的n级大数据格式所对应的切片信息以及三维局部块尺寸,可作为优选的实施方案,计算方式具体为:
(1)计算1级大数据格式对应的三维小块高和宽分别为h1、w1,计算方式为:
(2)计算1级大数据格式对应的每帧二维图像在x、y方向切片数量nx0和ny0,计算方式为:
其中,
(3)假设每一切片的x、y方向序号分别为idx1、idy1,idx1为x方向第idx1+1块,idy1为y方向第idy1+1块,则当前切片起点坐标为startPx1、startPy1,终点坐标为endPx1、endPy1,计算公式如下:
其中,min()为最小值。
(4)其它级大数据格式同1级大数据格式没有冗余,以相同的参数计算方式计算对应的切片信息以及三维局部块尺寸。
可作为优选的实施方案,上述利用每个三维小块z轴方向连续变化的信息,对该三维小块进行压缩,具体实现方式为:
通过三维小块图像数据的z轴方向连续性特征,使用三维小块中第一帧加上第二帧与第一帧的变化信息表示第二帧,第二帧加上第三帧与第二帧的变化信息表示第三帧,以此类推,实现对三维小块的压缩。
进一步可作为优选的实施方案,在对当前级大数据格式对应的各三维小块进行逐帧压缩的同时,每集齐三维局部块中连续两帧二维图像,即进行最大值采样,最终生成下一级大数据格式对应的三维局部块。
为了更好地说明本发明,现给出执行多级大数据格式归档的具体步骤,如下:
S01、设置0级大数据格式三维小块尺寸、0级大数据格式三维小块冗余尺寸参数信息,根据设置参数,计算大数据格式各级三维小块尺寸、原始图像序列切片信息。
具体的,在一具体实施例中,在步骤S01中,根据z轴连续性压缩方法,可快速解压读取任一帧图像信息,计算大数据格式切片信息时,默认z轴无冗余,且三维小块z轴长度为大数据图像序列长度。大数据格式仅0级支持冗余,其他级别用来可视化,无需支持冗余,故0级大数据格式三维小块尺寸与其他级别三维块尺寸不同,计算切块信息方式也不同,具体计算方式如下:
假设TB级图像序列长宽为bh、bw,序列长度为bz,0级大数据格式长宽为h0、w0,因本发明的压缩算法可快速直接读取任意帧,故默认0级三维小块z轴长度为图像序列长度bz,且z方向不设冗余,冗余尺寸为rh、rw。
S11、在大数据图像序列上0级大数据格式x、y方向切片数量分别为nx0、ny0,计算方式如下:
其中
S12、假设每一切片的x、y方向序号分别为idx0、idy0,idx0为x方向第idx0+1块,idy0为y方向第idy0+1块,则当前切片起点坐标为startPx0、startPy0,终点坐标为endPx0、endPy0,计算公式如下:
startPx0=(w0-rw)*idx0
startPy0=(h0-rh)*idy0
endPx0=min(startPx+w0,bw)
endPy0=min(startPy+h0,bh)
其中min()为最小值。
S13、局部块尺寸为sw、sh,因一个局部块需进行二级编码,一个二级块编码在x、y方向上需要4个0级块合成,直接读取局部块按行读取为连续读取,故局部块尺寸计算方式如下:
sw=bw
sh=3*(h0-w0)+h0
S14、1级大数据格式、2级大数据格式没有冗余,参数计算方式相同,以1级大数据格式参数计算方式为例。
1级大数据格式的尺寸分别为h1、w1,计算方式如下:
w1=w0-rw
h1=h0-rh
S15、在TB级图像序列上1级大数据格式x、y方向切片数量分别为nx1、ny1,计算方式如下:
其中
S16、假设每一切片的x、y方向序号分别为idx1、idy1,idx1为x方向第idx1+1块,idy1为y方向第idy1+1块,则当前切片起点坐标为startPx1、startPy1,终点坐标为endPx1、endPy1,计算公式如下:
startPx1=w1*idx1
startPy1=h1*idy1
endPx1=min(startPx+w1,bw)
endPy1=min(startPy+h1,bh)
其中min()为最小值。
S02、直接读取原始图像序列每一帧的局部切片,叠加每一帧读取的局部切片,得到对应的局部块,并创建0级大数据格式、1级大数据格式、2级大数据格式对应的存储容器。
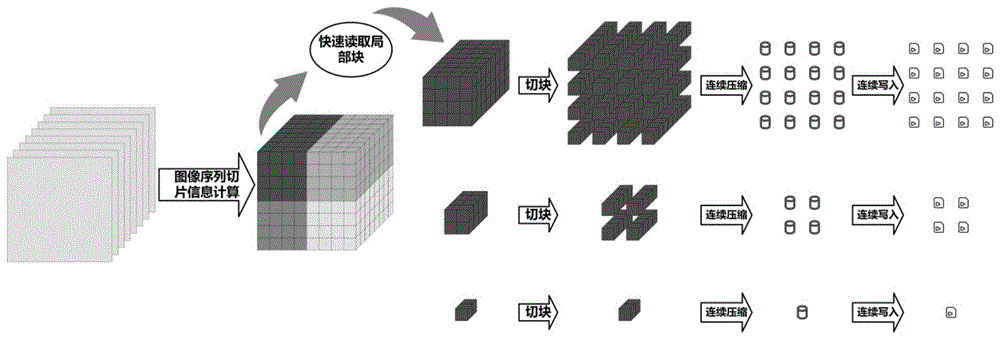
在该步骤S02中,读取局部数据,为直接连续读取磁盘中的局部图像数据,速度很快,且支持对压缩后的图像进行快速局部数据读取。直接快速读取局部数据,对每一帧,压缩后直接写入对应的存储容器中,这一过程对于IO操作为连续写入,磁盘利用率最高,写入速度快且稳定;其次无论三维图像序列尺寸如何,都可把原始图像划分为一个个完全独立处理的局部块,使其适用于所有尺寸的图像序列的大数据格式制作,局部块尺寸不大,相对于直接处理原始图像,IO稳定性强,连续写入操作多,写入速度快。
S03、根据S01中计算的大数据格式信息,对读取的局部块的每一帧切片,得到0级三维小块,0级三维小块是三维局部块每一帧xy面切片成若干个二维小片,叠加z方向每一帧相同xy位置的二维小块,得到0级三维小块。对每一个0级小块,利用原始图像序列z轴连续变化特点,进行压缩,并连续写入0级大数据格式对应的存储容器中。
也就是,在步骤S03中,对S02中读取到内存的局部块,进行0级大数据格式切块得到0级小块,对每一个0级小块利用z轴连续性压缩,使用第一帧加上第二帧与第一帧的变化信息表示第二帧,第二加上第三帧与第二帧的变化信息表示第三帧,依次下去,此方法可以表示任意一帧,使用此方法压缩三维图像并写入到0级大数据格式对应的idx_idy.avi存储容器中。其中0级大数据格式切片支持冗余切片,即连续两块三维小块数据允许存在重叠部分,便于后续全局分析。
在步骤S03中,0级大数据格式制作,支持冗余制作,即连续两块三维小块数据允许存在重叠部分,便于后续全局分析。
S04、集齐连续两帧局部块时,对此局部块进行x、y、z三个方向分别进行最大值采样,采样步长为2,得到1级局部块;对1级局部块的每一帧切片,得到1级三维小块,1级三维小块是三维局部块每一帧xy面切片成若干个二维小片,叠加z方向每一帧相同xy位置的二维小块,得到1级三维小块。对每一个1级小块,利用原始图像序列z轴连续变化特点,进行压缩,并连续写入1级大数据格式对应的存储容器中。
在该步骤S04中,集齐连续两帧S02中读取到内存的局部块,x、y、z三个方向分别进行最大值采样,采样步长为2,得到1级小块,对每一个1级小块利用z轴连续性压缩,使用第一帧加上第二帧与第一帧的变化信息表示第二帧,第二加上第三帧与第二帧的变化信息表示第三帧,依次下去,此方法可以表示任意一帧,使用此方法压缩三维图像并写入到1级大数据格式对应的idx_idy.avi存储容器中。
S05、集齐连续的2帧1级局部块时,对此局部块进行x、y、z三个方向分别进行最大值采样,采样步长为2,得到2级局部块;对2级局部块的每一帧切片,得到2级三维小块,2级三维小块是三维局部块每一帧xy面切片成若干个二维小片,叠加z方向每一帧相同xy位置的二维小块,得到2级三维小块。对每一个2级小块,利用原始图像序列z轴连续变化特点,进行压缩,并连续写入2级大数据格式对应的存储容器中。
在步骤S05中,集齐连续两帧S04中1级小块,x、y、z三个方向分别进行最大值采样,采样步长为2,得到2级小块,对每一个2级小块利用z轴连续性压缩,使用第一帧加上第二帧与第一帧的变化信息表示第二帧,第二加上第三帧与第二帧的变化信息表示第三帧,依次下去,此方法可以表示任意一帧,使用此方法压缩三维图像并写入到级大数据格式对应的idx_idy.avi存储容器中。
S06、每一帧局部块都按照上述处理完后,大数据格式制作完成,上述方法只可得到0级、1级、2级3层大数据格式。若需要更多级的大数据格式,可直接对2级大数据格式进行采样合并处理,因2级大数据格式的大小已比原始图像小100倍以上,故不用考虑性能问题。
在步骤S06中,步骤S01中的所有局部块,按照S02~S05中所有步骤处理完毕,大数据格式制作完成。
需要说明的是,在步骤S03、S04、S05中的压缩算法,通过生物医学三维图像数据的z轴连续性特征,使用第一帧加上第二帧与第一帧的变化信息表示第二帧,第二帧加上第三帧与第二帧的变化信息表示第三帧,依次下去,此方法可以表示任意一帧,使用此方法压缩三维图像,压缩速度极快、压缩比高、压缩图像解码速度快,以实现大数据格式的快速制作。
另外,本实施例方法在读取三维局部块时,相对于传统方法一帧分为多个部分,每次读取一部分,然后解码,再读另一部分,当并行读图时,会有大量的随机读取,对于硬盘随机读取性能很低,读图速度慢。本实施例方法提出采用依次串行读取三维局部块的每一帧,同时并行解码读取的每一帧数据的方法,极大的降低了读图时随机读取的次数,理论上达到硬盘读取速度极限。
总的来说,本实施例方法将生物医学三维图像z轴的连续性,用于三维图像的压缩,同时提出直接快速读取局部图像方法,实现TB级生物医学三维图像数据大数据格式的快速制作。具体地,计算大数据图像的切块信息;直接快速读取局部块,无论三维图像序列尺寸如何,都可把原始图像划分为一个个完全独立处理的局部块,使其适用于所有尺寸的TB级生物医学三维图像数据大数据格式的制作,其次局部块尺寸不大,相对于直接处理原始图像,IO稳定性强,连续写入操作多,写入速度快;在内存中结合计算的切块信息对局部块进行切片,最后利用z轴连续性特征对图像进行压缩并连续写入对应的大数据格式的存储容器中,实现TB级生物医学三维图像数据大数据格式的快速制作。该方法制作速度快,压缩比高,并且解压缩即还原图像速度也很快,稳定性强,可解决现有技术制作存在的高成本、易用性差、实用性低等问题。
下面结合附图来对本发明的方法做进一步详细说明:
如图2所示,本发明TB级生物医学三维图像数据大数据格式的制作方法,计算TB级图像序列切片信息,将图像序列划分为若干三维局部块,三维局部块是图像序列每一帧xy面切片成若干个二维小片,叠加z方向每一帧相同xy位置的二维小块,得到三维局部块;直接快速读取一个三维局部块;对三维局部块切片,得到0级三维小块,0级三维小块是三维局部块z轴每一帧xy面切片成若干个二维小片,叠加z方向每一帧相同xy位置的二维小块,得到若干个0级三维小块;对每一个0级小块利用z轴连续性压缩,使用第一帧加上第二帧与第一帧的变化信息表示第二帧,第二加上第三帧与第二帧的变化信息表示第三帧,依次下去,此方法可以表示任意一帧,使用此方法连续压缩,得到若干个压缩后的0级三维小块;压缩后的0级三维小块是一个整体,将压缩后的0级三维小块连续写入到对应的0级大数据格式存储容器中;再对三维局部块进行下采样,得到1级三维局部块,和0级大数据格式制作类似,对1级三维局部块切片、压缩并连续写入到对应的1级大数据格式存储容器中;对1级三维局部块下采样,类似的方法切片、压缩并连续写入到对应的2级大数据格式存储容器中;所有局部块处理完,大数据格式制作完成。
实施例二
一种计算机可读存储介质,所述计算机可读存储介质包括存储的计算机程序,其中,在所述计算机程序被处理器运行时控制所述存储介质所在设备执行如上所述的一种TB级生物医学三维图像数据的大数据格式归档方法。
相关技术方案同实施例一,在此不再赘述。
本领域的技术人员容易理解,以上所述仅为本发明的较佳实施例而已,并不用以限制本发明,凡在本发明的精神和原则之内所作的任何修改、等同替换和改进等,均应包含在本发明的保护范围之内。
- 一种基于格式化数据的数据格式校查系统及其方法
- 一种大数据归档方法、装置、设备及存储介质
- 一种基于环保大数据的分类归档方法
