DNA扩增反应中的不可延伸的寡核苷酸
文献发布时间:2024-04-18 20:01:23
对相关申请的引用
本申请要求2021年3月2日提交的美国临时申请号63/155,398和2020年5月27日提交的美国临时申请号63/030,452的优先权,所述美国临时申请中的每一个的全部内容通过引用并入本文。
关于联邦资助研究的声明
本发明是在美国国立卫生研究院授予的授权号R01CA203964下在政府资助的情况下完成的。政府享有本发明中的某些权利。
对序列表的引用
本申请含有已经以ASCII格式经由EFS-Web递交的序列表并且所述序列表据此通过引用整体并入。于2021年5月18日创建的所述ASCII副本被命名为RICEP0076WO_ST25.txt并且大小为55.3千字节。
背景技术
此公开的开发部分在授权号RP180147下由德克萨斯州癌症预防和研究所(CPRIT)资助。
1.技术领域
本发明总体上涉及分子生物学领域。更具体地说,本发明涉及包括不可延伸的寡核苷酸(NEO)的组合物和使用该不可延伸的寡核苷酸的方法,该不可延伸的寡核苷酸在其3′端处或附近具有合理设计的二级结构,其不被具有3′->5′核酸外切酶活性的高保真度DNA聚合酶酶促延伸。
2.相关技术描述
DNA聚合酶用于各种应用中,以延伸DNA寡核苷酸。然而,存在某些情况,其中希望含有DNA聚合酶的溶液中的某些DNA寡核苷酸不被酶促延伸。历史上,研究人员已经使用在3′端具有化学修饰的DNA寡核苷酸来防止酶促的延伸;这些修饰包含反向DNA核苷酸、聚乙二醇间隔子、基于烷烃的间隔子、荧光团、猝灭剂、小沟结合剂等。这些化学修饰在寡核苷酸合成后对DNA寡核苷酸进行功能化是昂贵的,显著降低了寡核苷酸的纯度和产量,并显著延长了合成周转时间。由于这些原因,需要未被化学修饰的不可延伸的寡核苷酸。
发明内容
因此,本文提供了不可延伸的寡核苷酸(NEO),其通过合理设计的NEO的3′端处的二级结构抑制酶促延伸。
在一个实施例中,本文提供了组合物,所述组合物包括DNA模板、DNA聚合酶和不可延伸的寡核苷酸,其中所述DNA模板从5′至3′连续地包括上游序列和探针结合序列,其中所述不可延伸的寡核苷酸从5′至3′包括:结合序列,所述结合序列与所述DNA模板的所述探针结合序列的反向互补序列至少70%相同,终止子发夹,所述终止子发夹位于不可延伸的寡核苷酸的3′端,所述终止子发夹包括:第一茎序列,第二茎序列,其中所述第二茎序列是所述第一茎序列的反向互补序列,以及环序列,所述环序列位于所述第一茎序列和所述第二茎序列之间。例如,所述结合序列与所述DNA模板的所述探针结合序列的反向互补序列至少70%、75%、80%、85%、90%、95%、96%、97%、98%或99%相同。
在一些方面,所述不可延伸的寡核苷酸的所述结合序列为10至300个核苷酸长。例如,所述不可延伸的寡核苷酸的所述结合序列可以为10-300、10-250、10-200、10-150、10-100、10-90、10-80、10-70、10-60、10-50、10-45、10-40、10-35、10-30、15-30、15-35、15-40、15-45、15-50、20-35、20-40、20-45或20-50个核苷酸长。例如,所述不可延伸的寡核苷酸的所述结合序列可以为至少或约10、15、20、25、30、35、40、45、50、55、60、65、70、75、80、85、90、95、100、120、140、160、180、200、220、240、260、280或300个核苷酸长。
在一些方面,所述不可延伸的寡核苷酸的所述终止子发夹不是所述DNA模板的所述上游序列的反向互补序列。在一些方面,所述不可延伸的寡核苷酸的所述终止子发夹不能与所述DNA模板的所述上游序列杂交。
在一些方面,所述终止子发夹的所述第一茎序列为3至8个核苷酸长。例如,所述终止子发夹的所述第一茎序列可以为至少或约3、4、5、6、7或8个核苷酸长。在一些方面,所述终止子发夹的所述第一茎序列为四个核苷酸长。在一些方面,所述终止子发夹的所述第二茎序列为3至8个核苷酸长。例如,所述终止子发夹的所述第二茎序列可以为至少或约3、4、5、6、7或8个核苷酸长。在某些方面,所述终止子发夹的所述第二茎序列为四个核苷酸长。在一些方面,所述终止子发夹的所述第一茎序列和所述第二茎序列均为四个核苷酸长。
在一些方面,所述终止子发夹具有腺嘌呤核苷酸作为其最3′-核苷酸。在一些方面,所述第一茎序列为5′-TCTC-3′,并且所述第二茎序列为5′-GAGA-3′。在一些方面,所述第一茎序列为5′-GTTC-3′,并且所述第二茎序列为5′-GAAC-3′。
在一些方面,所述终止子发夹的所述环序列为3至10个核苷酸长。例如,所述终止子发夹的所述环序列可以为至少或约3、4、5、6、7、8、9或10个核苷酸长。在一些方面,所述终止子发夹的所述环序列为四个核苷酸长。在一些方面,所述环序列为5′-GCAA-3′。
在一些方面,所述不可延伸的寡核苷酸进一步包括位于所述结合序列和所述终止子发夹之间的中间发夹。所述中间发夹可以包括第三茎序列、第四茎序列和第二环序列,所述第二环序列位于所述第三茎序列和所述第四茎序列之间,其中所述第四茎序列是所述第三茎序列的反向互补序列。在一些方面,所述终止子发夹的最3′-核苷酸是胞嘧啶。在一些方面,所述终止子发夹的所述第一茎序列和所述终止子发夹的所述第二茎序列各自为3至8个核苷酸长。例如,所述终止子发夹的所述第一茎序列和所述第二茎序列可以各自独立地为至少或约3、4、5、6、7或8个核苷酸长。在一些方面,所述第一茎序列为5′-GTTA-3′,并且所述第二茎序列为5′-TAAC-3′。在一些方面,所述第一茎序列为5′-GATT-3′,并且所述第二茎序列为5′-AATC-3′。在一些方面,所述终止子发夹的所述第一环序列为3至10个核苷酸长。例如,所述第一环序列可以为至少或约3、4、5、6、7、8、9或10个核苷酸长。在一些方面,所述第一环序列为5′-GCAA-3′。在一些方面,所述中间发夹的所述第三茎序列和所述中间发夹的所述第四茎序列各自为3至20个核苷酸长。例如,所述中间发夹的所述第三茎序列和所述第四茎序列可以各自独立地为至少或约3、4、5、6、7、8、9、10、11、12、13、14、15、16、17、17、19或20个核苷酸长。在一些方面,所述第三茎序列为5′-GAGAAC-3′,并且所述第四茎序列为5′-GTTCTC-3′。在一些方面,所述第三茎序列为5′-CCTGTA-3′,并且所述第四茎序列为5′-TACAGG-3′。在一些方面,所述中间发夹的所述第二环序列为3至15个核苷酸长。例如,所述中间发夹的所述第二环序列可以为至少或约3、4、5、6、7、8、9、10、11、12、13、14或15个核苷酸长。在一些方面,所述中间发夹的所述第二环序列为5′-ATTA-3′。在一些方面,所述中间发夹的所述第二环序列为5′-CACA-3′。在某些方面,所述第一茎序列为5′-GTTA-3′,所述第二茎序列为5′-TAAC-3′,所述第一环序列为5′-GCAA-3′,所述第三茎序列为5′-GAGAAC-3′,所述第四茎序列为5′-GTTCTC-3′,并且所述中间发夹的所述第二环序列为5′-ATTA-3′。在某些方面,所述第一茎序列为5′-GATT-3′,所述第二茎序列为5′-AATC-3′,所述第一环序列为5′-GCAA-3′,所述第三茎序列为5′-GAGAAC-3′,所述第四茎序列为5′-GTTCTC-3′,并且所述中间发夹的所述第二环序列为5′-ATTA-3′。在某些方面,所述第一茎序列为5′-GTTA-3′,所述第二茎序列为5′-TAAC-3′,所述第一环序列为5′-GCAA-3′,所述第三茎序列为5′-CCTGTA-3′,所述第四茎序列为5′-GGACAT-3′,并且所述中间发夹的所述第二环序列为5′-CACA-3′。
在一些方面,所述不可延伸的寡核苷酸进一步包括位于所述结合序列和所述终止子发夹之间的错配序列。在一些方面,所述错配序列为1至100个核苷酸长。例如,所述错配序列可以为1-100、1-90、1-80、1-70、1-60、1-50、1-40、1-30、1-25、1-20、1-15、1-10、5-10、5-15、5-20、10-20、10-25或10-30个核苷酸长。例如,所述错配序列可以为至少或约1、2、3、4、5、6、7、8、9、10、15、20、25、30、35、40、45、50、55、60、65、70、75、80、85、90、95或100个核苷酸长。在一些方面,所述错配序列与所述DNA模板的所述上游序列的反向互补序列至多30%相同。例如,所述错配序列可以与所述DNA模板的所述上游序列的反向互补序列至多或约5%、10%、15%、20%、25%或30%相同。在一些方面,所述错配序列不能与所述DNA模板的所述上游序列杂交。
在一些方面,所述错配序列不形成非线性二级结构。在一些方面,对于比-2kcal/mol更强的分子内结构,不存在所述错配序列的两个子序列。在一些方面,所述错配序列不形成发夹。在一些方面,所述错配序列为5至20个核苷酸长。例如,所述错配序列为至少或约5、6、7、8、9、10、11、12、13、14、15、16、17、18、19或20个核苷酸长。
在一些方面,所述错配序列包括前一子序列和后一子序列,其中所述后一子序列是所述前一子序列的反向互补序列。在一些方面,所述前一子序列和所述后一子序列各自为至少四个核苷酸长。在一些方面,所述前一子序列和所述后一子序列各自为六个核苷酸长。在一些方面,所述错配序列包括多个前一子序列和多个后一子序列,其中每个前一子序列是相应后一子序列的反向互补序列。在一些方面,每个前一子序列和每个后一子序列为至少四个核苷酸长。
在一些方面,所述错配序列从5′至3′包括第一子序列、第二子序列、第三子序列和第四子序列,其中所述第一子序列是所述第二子序列的反向互补序列,并且其中所述第三子序列是所述第四子序列的反向互补序列。在一些方面,所述第一子序列、所述第二子序列、所述第三子序列和所述第四子序列中的每一个为四至15个核苷酸长。例如,所述第一子序列、所述第二子序列、所述第三子序列和所述第四子序列中的每一个可以为至少或约4、5、6、7、8、9、10、11、12、13、14或15个核苷酸长。
在一些方面,所述错配序列从5′至3′包括第一子序列、第二子序列、第三子序列和第四子序列,其中所述第一子序列是所述第四子序列的反向互补序列,并且其中所述第二子序列是所述第三子序列的反向互补序列。在一些方面,所述第一子序列、所述第二子序列、所述第三子序列和所述第四子序列中的每一个为四至15个核苷酸长。例如,所述第一子序列、所述第二子序列、所述第三子序列和所述第四子序列中的每一个可以为至少或约4、5、6、7、8、9、10、11、12、13、14或15个核苷酸长。在一些方面,非互补区域位于由所述第一子序列和所述第四子序列形成的所述双链区域与由所述第二子序列和所述第三子序列形成的所述双链区域之间。在一些方面,所述非互补区域为3至10个核苷酸长。例如,所述非互补区域可以为至少或约3、4、5、6、7、8、9或10个核苷酸长。
在一些方面,所述不可延伸的寡核苷酸在其3′端不包括人工化学修饰或非天然的DNA核苷酸。
在一些方面,所述DNA模板的所述上游序列为3至100个核苷酸长。例如,所述DNA模板的所述上游序列可以为3-100、3-90、3-80、3-70、3-60、3-50、3-40、3-30、3-25、3-20、5-15、5-20、5-25、5-30、5-35、10-20、10-25或10-30个核苷酸长。例如,所述DNA模板的所述上游序列可以为至少或约3、4、5、6、7、8、9、10、15、20、25、30、35、40、45、50、55、60、65、70、75、80、85、90、95或100个核苷酸长。
在一些方面,所述DNA模板的所述探针结合序列为10至300个核苷酸长。例如,所述DNA模板的所述探针结合序列可以为10-300、10-250、10-200、10-150、10-100、10-90、10-80、10-70、10-60、10-50、10-45、10-40、10-35、10-30、15-30、15-35、15-40、15-45、15-50、20-35、20-40、20-45或20-50个核苷酸长。例如,所述DNA模板的所述探针结合序列可以为至少或约10、15、20、25、30、35、40、45、50、55、60、65、70、75、80、85、90、95、100、120、140、160、180、200、220、240、260、280或300个核苷酸长。
在一些方面,所述DNA聚合酶是具有3′至5′核酸外切酶活性的高保真度DNA聚合酶。
在一些方面,所述组合物可以包括不可延伸的寡核苷酸的群体和DNA模板的群体,其中所述群体的各种不可延伸的寡核苷酸具有不同的结合序列,这些结合序列与在DNA模板的群体中发现的各种探针结合序列的反向互补序列至少70%相同。例如,所述组合物可以包括至少或约2、3、4、5、6、7、8、9、10、11、12、13、14、15或20种不同的不可延伸的寡核苷酸。
在一个实施例中,本文提供了包括DNA模板、DNA聚合酶和不可延伸的寡核苷酸的组合物,其中所述DNA模板从5′至3′连续地包括上游序列和探针结合序列,其中所述不可延伸的寡核苷酸从5′至3′包括:结合序列,所述结合序列与所述DNA模板的所述探针结合序列的反向互补序列至少70%相同;包括第一茎序列和第二茎序列的错配序列,其中所述第二茎序列是所述第一茎序列的反向互补序列;以及尾部序列,所述尾部序列与所述DNA模板的所述上游序列的反向互补序列至多40%相同。例如,所述结合序列与所述DNA模板的所述探针结合序列的反向互补序列至少70%、75%、80%、85%、90%、95%、96%、97%、98%或99%相同。例如,所述尾部序列与所述DNA模板的所述上游序列的反向互补序列至多或约5%、10%、15%、20%、25%、30%、35%或40%相同。
在一些方面,所述不可延伸的寡核苷酸的所述结合序列为10至300个核苷酸长。例如,所述不可延伸的寡核苷酸的所述结合序列可以为10-300、10-250、10-200、10-150、10-100、10-90、10-80、10-70、10-60、10-50、10-45、10-40、10-35、10-30、15-30、15-35、15-40、15-45、15-50、20-35、20-40、20-45或20-50个核苷酸长。例如,所述不可延伸的寡核苷酸的所述结合序列可以为至少或约10、15、20、25、30、35、40、45、50、55、60、65、70、75、80、85、90、95、100、120、140、160、180、200、220、240、260、280或300个核苷酸长。
在一些方面,所述不可延伸的寡核苷酸的所述错配序列为10至100个核苷酸长。例如,所述错配序列可以为10-100、10-90、10-80、10-70、10-60、10-50、10-40、10-30、10-25或10-20个核苷酸长。例如,所述错配序列可以为至少或约10、11、12、13、14、15、16、17、18、19、20、25、30、35、40、45、50、55、60、65、70、75、80、85、90、95或100个核苷酸长。
在一些方面,所述错配序列的所述第一茎序列为4至45个核苷酸长。例如,所述错配序列的所述第一茎序列可以为4-45、4-40、4-35、4-30、4-25、4-20、4-15、4-10、8-45、8-40、8-35、8-30、8-25、8-20或8-15个核苷酸长。例如,所述错配序列的所述第一茎序列可以为至少或约4、5、6、7、8、9、10、11、2、13、14、15、20、25、30、35、40或45个核苷酸长。在一些方面,所述错配序列的所述第二茎序列为4至45个核苷酸长。例如,所述错配序列的所述第二茎序列可以为4-45、4-40、4-35、4-30、4-25、4-20、4-15、4-10、8-45、8-40、8-35、8-30、8-25、8-20或8-15个核苷酸长。例如,所述错配序列的所述第二茎序列可以为至少或约4、5、6、7、8、9、10、11、2、13、14、15、20、25、30、35、40或45个核苷酸长。
在一些方面,所述错配序列包括多个第一茎序列和多个第二茎序列,其中每个第二茎序列是相应的第一茎序列的反向互补序列。在一些方面,每个第一茎序列和每个第二茎序列为四至45个核苷酸长。例如,每个第一茎序列和每个第二茎序列可以为4-45、4-40、4-35、4-30、4-25、4-20、4-15、4-10、8-45、8-40、8-35、8-30、8-25、8-20或8-15个核苷酸长。例如,每个第一茎序列和每个第二茎序列可以为至少或约4、5、6、7、8、9、10、11、2、13、14、15、20、25、30、35、40或45个核苷酸长。
在一些方面,所述错配序列从5′至3′包括第一子序列、第二子序列、第三子序列和第四子序列,其中所述第一子序列是所述第二子序列的反向互补序列,并且其中所述第三子序列是所述第四子序列的反向互补序列。在一些方面,所述第一子序列、所述第二子序列、所述第三子序列和所述第四子序列中的每一个为四至15个核苷酸长。例如,所述第一子序列、所述第二子序列、所述第三子序列和所述第四子序列中的每一个可以为至少或约4、5、6、7、8、9、10、11、12、13、14或15个核苷酸长。
在一些方面,所述错配序列从5′至3′包括第一子序列、第二子序列、第三子序列和第四子序列,其中所述第一子序列是所述第四子序列的反向互补序列,并且其中所述第二子序列是所述第三子序列的反向互补序列。在一些方面,所述第一子序列、所述第二子序列、所述第三子序列和所述第四子序列中的每一个为四至15个核苷酸长。例如,所述第一子序列、所述第二子序列、所述第三子序列和所述第四子序列中的每一个可以为至少或约4、5、6、7、8、9、10、11、12、13、14或15个核苷酸长。在一些方面,非互补区域位于由所述第一子序列和所述第四子序列形成的所述双链区域与由所述第二子序列和所述第三子序列形成的所述双链区域之间。在一些方面,所述非互补区域为3至10个核苷酸长。例如,所述非互补区域可以为至少或约3、4、5、6、7、8、9或10个核苷酸长。
在一些方面,所述尾部序列为3至15个核苷酸长。例如,所述尾部序列可以为至少或约3、4、5、6、7、8、19、10、11、12、13、14或15个核苷酸长。在一些方面,所述不可延伸的寡核苷酸的所述尾部序列不能与所述DNA模板的所述上游序列杂交。在一些方面,所述不可延伸的寡核苷酸的所述尾部序列不形成非线性二级结构。在一些方面,所述不可延伸的寡核苷酸的所述尾部序列不形成发夹。
在一些方面,所述不可延伸的寡核苷酸在其3′端不包括人工化学修饰或非天然的DNA核苷酸。
在一些方面,所述DNA模板的所述上游序列为3至100个核苷酸长。例如,所述DNA模板的所述上游序列可以为3-100、3-90、3-80、3-70、3-60、3-50、3-40、3-30、3-25、3-20、5-15、5-20、5-25、5-30、5-35、10-20、10-25或10-30个核苷酸长。例如,所述DNA模板的所述上游序列可以为至少或约3、4、5、6、7、8、9、10、15、20、25、30、35、40、45、50、55、60、65、70、75、80、85、90、95或100个核苷酸长。
在一些方面,所述DNA模板的所述探针结合序列为10至300个核苷酸长。例如,所述DNA模板的所述探针结合序列可以为10-300、10-250、10-200、10-150、10-100、10-90、10-80、10-70、10-60、10-50、10-45、10-40、10-35、10-30、15-30、15-35、15-40、15-45、15-50、20-35、20-40、20-45或20-50个核苷酸长。例如,所述DNA模板的所述探针结合序列可以为至少或约10、15、20、25、30、35、40、45、50、55、60、65、70、75、80、85、90、95、100、120、140、160、180、200、220、240、260、280或300个核苷酸长。
在一些方面,所述DNA聚合酶是具有3′至5′核酸外切酶活性的高保真度DNA聚合酶。
在一些方面,所述组合物可以包括不可延伸的寡核苷酸的群体和DNA模板的群体,其中所述群体的各种不可延伸的寡核苷酸具有不同的结合序列,这些结合序列与在DNA模板的群体中发现的各种探针结合序列的反向互补序列至少70%相同。例如,所述组合物可以包括至少或约2、3、4、5、6、7、8、9、10、11、12、13、14、15或20种不同的不可延伸的寡核苷酸。
在一个实施例中,本文提供了用于选择性地抑制具有选定序列的模板DNA的聚合酶链式反应(PCR)扩增的方法,所述方法包括:(a)在适合于DNA聚合酶活性的条件下,混合本实施例中的任何一种的组合物、正向引物、反向引物和dNTP;以及(b)使所述混合物经受至少7轮热循环。例如,所述热循环可以进行至少或约7、8、9、10、11、12、13、14、15、16、17、18、19、20、25、30、35或40个循环。
在一些方面,每轮热循环包括首先将所述混合物在至少78℃的温度下保持1秒至30分钟,然后将所述混合物在至多75℃的温度下保持1秒至4小时。例如,第一步骤可以包括将混合物保持在至少78℃、79℃、80℃、81℃、82℃、83℃、84℃、85℃、86℃、87℃、88℃、89℃、90℃、91℃、92℃、93℃、94℃、95℃、96℃、97℃或98℃的温度下。例如,第一步骤可以包括在该温度下保持1秒至30分钟、10秒至30分钟、20秒至30分钟、30秒至30分钟、45秒至30分钟、1分钟至30分钟、2分钟至30分钟、30秒至5分钟或1分钟至5分钟。例如,第一步骤可以包括在该温度下保持至少或约1秒、5秒、10秒、15秒、20秒、30秒、45秒、1分钟、2分钟、5分钟或10分钟。例如,第二步骤可以包括将混合物保持在至多75℃、74℃、73℃、72℃、71℃、70℃、69℃、68℃、67℃、66℃、65℃、64℃、63℃、62℃、61℃、60℃、59℃、58℃、57℃、56℃或55℃的温度下。例如,第二步骤可以包括在该温度下保持1秒至4小时、1秒至3小时、1秒至2小时、1秒至1小时、1秒至30分钟、10秒至30分钟、20秒至30分钟、30秒至30分钟、45秒至30分钟、1分钟至30分钟、2分钟至30分钟、30秒至5分钟或1分钟至5分钟。例如,第一步骤可以包括在该温度下保持至少或约1秒、5秒、10秒、15秒、20秒、30秒、45秒、1分钟、2分钟、5分钟或10分钟。
在一些方面,所述正向引物为12至60个核苷酸长。在一些方面,所述正向引物与所述DNA模板的子序列的反向互补序列至少80%、85%、90%、95%、96%、97%、98%或99%相同。在一些方面,所述反向引物为12至60个核苷酸长。在一些方面,所述反向引物与所述DNA模板的子序列至少80%、85%、90%、95%、96%、97%、98%或99%相同。
在一些方面,所述DNA模板任选地包括靶DNA模板。在一些方面,所述DNA模板包括背景DNA模板。
在一些方面,所述不可延伸的寡核苷酸具有与所述背景DNA模板的所述探针结合序列的反向互补序列至少80%、85%、90%、95%、96%、97%、98%或99%同源的结合序列。在一些方面,所述不可延伸的寡核苷酸在其3′端不包括人工化学修饰或非天然的DNA核苷酸。
在一些方面,所述背景DNA模板是假基因。在一些方面,所述靶DNA模板是与所述假基因具有高于80%、85%、90%、95%、96%、97%、98%或99%同源性的基因序列。在一些方面,背景DNA模板是野生型基因序列。在一些方面,所述靶DNA模板是具有单个核苷酸替换、两个核苷酸替换、1至50个核苷酸的插入或1至50个核苷酸的缺失的变体基因序列。例如,插入或缺失可以为1、2、3、4、5、6、7、8、9、10、11、12、13、14、15、20、25、30、35、40、45或50个核苷酸长。
在一些方面,步骤(a)使用本实施例的T1NEO中的任何一种的组合物进行。在一些方面,步骤(a)使用本实施例的T2NEO中的任何一种的组合物进行。
在一些方面,所述不可延伸的寡核苷酸的所述结合序列与所述正向引物的15个核苷酸子序列至少70%、75%、80%、85%、90%、95%、96%、97%、98%或99%同源。在一些方面,步骤(a)的所述混合物包括100pM至5μM的所述正向引物,100pM至5μM的所述反向引物,以及100pM至5μM的所述不可延伸的寡核苷酸。例如,所述混合物中所述正向引物、反向引物和不可延伸的寡核苷酸中的任何一种的浓度独立地可以为100pM-5μM、200pM-5μM、300pM-5μM、400pM-5μM、500pM-5μM、750pM-5μM、1nM-5μM、250nM-5μM、500nM-5μM、750nM-5μM、1μM-5μM、100pM-1μM、200pM-1μM、300pM-1μM、400pM-1μM、500pM-1μM、750pM-1μM、1nM-1μM或500pM-500nM。例如,所述混合物中所述正向引物、反向引物和不可延伸的寡核苷酸中的任何一种的浓度独立地可以为至少或约100pM、200pM、300pM、400pM、500pM、750pM、1nM、10nM、50nM、100nM、200nM、300nM、400nM、500nM、750nM、1μM、2μM、3μM、4μM或5μM。
在一些方面,所述DNA聚合酶是具有3′至5′核酸外切酶活性的高保真度DNA聚合酶。
在一些方面,步骤(a)的所述混合物进一步包括嵌入染料DNA或Taqman探针。在一些方面,基于循环阈值(Ct)值确定所述靶DNA模板的量或浓度。
在一些方面,所述正向引物在其5′端进一步包括正向衔接子,并且所述反向引物在其5′端进一步包括反向衔接子,并且所述方法进一步包括(c)进行高通量测序。在一些方面,所述方法进一步包括(c)将衔接子序列连接到步骤(b)中产生的PCR产物,以及(d)进行高通量测序。
在一些方面,所述方法可以使用不可延伸的寡核苷酸的群体来进行,以扩增具有各种选定序列的DNA模板的群体,其中所述群体的各种不可延伸的寡核苷酸具有不同的结合序列,这些结合序列与在DNA模板的群体中发现的各种探针结合序列的反向互补序列至少70%相同。例如,可以使用至少或约2、3、4、5、6、7、8、9、10、11、12、13、14、15或20种不同的不可延伸的寡核苷酸来实施这些方法。
在一些方面,当方法需要定量PCR时,所述可以使用实例1中提供的反应方案和条件来进行。在一些方面,当方法需要下一代测序时,所述可以使用实例2中提供的反应方案和条件来进行。在一些方面,当方法需要NGS生物信息学分析时,所述可以使用实例3中提供的方法进行。在一些方面,当方法需要倍数富集分析或变体等位基因频率分析时,所述可以使用实例4中提供的方法进行。
本发明的其他目的、特征和优点将从以下详细描述中变得清楚。然而,应当理解,虽然详细说明和具体实例指示了本发明的优选实施例,但仅以说明的方式给出,因为在本发明的精神和范围内的各种变化和修改对于本领域技术人员来说将变得明显。
附图说明
以下附图形成本说明书的一部分,并且包含在内以进一步证明本发明的某些方面。通过参考这些附图中的一个或多个并结合本文呈现的具体实施例的详细描述,可以更好地理解本发明。
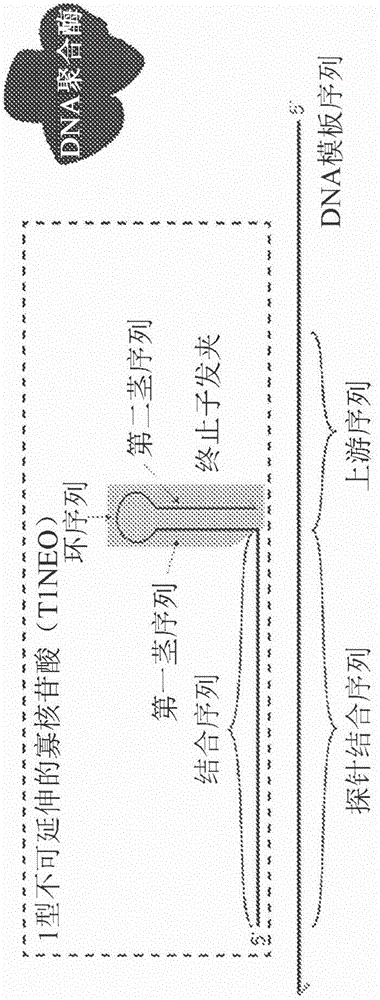
图1:本发明的关键成分包含1型不可延伸的寡核苷酸(T1NEO)。虚线框表示T1NEO。DNA模板从5′至3′连续地具有上游序列和探针结合序列。模板序列的左侧和T1NEO的右侧的灰色箭头表示寡核苷酸的3′端。T1NEO上的结合序列和模板上的探针结合序列大部分或完全反向互补。T1NEO的最3′区域的终止子发夹具有与第二茎序列反向互补的第一茎序列。终止子发夹在第一茎和第二茎序列之间具有环序列,如终止子发夹的顶部的弧线所示。该系统还包含DNA聚合酶。
图2:T1NEO的两个实施例。顶部的实施例具有终止子发夹序列5′-TCTCGCAAGAGA-3′(SEQ ID NO:244)。底部的实施例具有终止子发夹序列5′-GTTCGCAAGAAC-3′(SEQ ID NO:245)。
图3:在结合序列和终止子发夹之间具有错配序列(MS)的T1NEO。MS不与DNA模板的上游序列大部分或完全反向互补。
图4:具有包括发夹的MS的T1NEO。如图所示,MS包括具有反向互补序列的前一子序列和后一子序列。MS可以另外包括不形成发夹的附加子序列。
图5:具有包括堆叠的发夹或多个发夹的MS的T1NEO。在两个实施例中,T1NEO从5′至3′的顺序包括第一子序列、第二子序列、第三子序列和第四子序列。在顶部的实施例中,第一子序列与第四子序列反向互补,并且第二子序列与第三子序列反向互补,以形成堆叠的发夹。在底部的实施例中,第一子序列与第二子序列反向互补,并且第三子序列与第四子序列反向互补,以形成两个独立的发夹。
图6:具有10nt非结构化MS和包括序列5′-TCTCGCAAGAGA-3′(SEQ ID NO:244)的终止子发夹的T1NEO的实验证明。此处,使用具有3′->5′核酸外切酶活性的Phusion高保真度DNA聚合酶,并且使用Syto-13嵌入染料,将定量PCR(qPCR)应用于NA18537人类基因组DNA模板。在灰色线中,正向引物(FP;5′-GAGGGGTATTAGAAGAATGACTATGTGA-3′;SEQ ID NO:85)和反向引物(RP;5′-ACATGGTTAGATATTAGCCTGACCTATG-3′;SEQ ID NO:165)显示出能有效地进行qPCR扩增。相反,当FP被T1NEO(5′-GACTATGTGACAAAATAGCTAAGGATACAGGAAATATGGAACATTAGTTCTCGCAAGAGA-3′;SEQ ID NO:246)替换时,即使当使用10倍高浓度的T1NEO,也没有发生可观察到的PCR扩增。这些数据支持这样的假设,即T1NEO甚至不太可能被具有3′->5′核酸外切酶活性的DNA聚合酶酶促延伸。三条T1NEO迹线中的一个的晚期荧光增加可能是由于RP引物二聚体或基因组上的非特异性扩增。
图7:T1NEO的附加实施例的实验证明。顶部的实施例显示了没有MS的T1NEO(5′-GACTATGTGACAAAATAGCTAAGGATACAGGAAATATGTCTCGCAAGAGA-3′;SEQ ID NO:246)。中间的实施例显示了具有不同终止子发夹序列的T1NEO(5′-GACTATGTGACAAAATAGCTAAGGATACAGGAAATATGGAACATTAGTACTCGCAAGAGT-3′;SEQ ID NO:247)。底部的实施例显示了具有第三终止子发夹序列的T1NEO(5′-GACTATGTGACAAAATAGCTAAGGATACAGGAAATATGGAACATTAGTGTTCGCAAGAAC-3′;SEQ ID NO:248)。所有三个实施例在抑制DNA聚合酶延伸方面都是有效的,但是底部的实施例似乎最适合这个基因组基因座。
图8:在MS中包括多个发夹和堆叠的发夹的T1NEO的附加实施例的实验证明在此处显示的两个实施例(顶部:5′-GACTATGTGAcAAAATAGCTAAGGATACAGGAAATATGCGTAAGTCATCTTACGTGAGAGAACATTAGTTCTCCTTCTCCGTTGAGA-3′;SEQ ID NO:249;中间和底部:5′-GACTATGTGAcAAAATAGCTAAGGATACAGGAAATATGGAGAACATTACCTGTATGATACAGGCACAGTTCTCCTTCTCCGTTGAGA-3′;SEQ ID NO:250)表现出对该基因组基因座的DNA聚合酶延伸的近乎完美的抑制。使用100nM和1000nM T1NEO测试堆叠的发夹T1NEO;两个实验都显示了对Phusion DNA聚合酶延伸的近乎完美的抑制,证明了qPCR信号的缺乏不是由于来自高浓度T1NEO的非特异性抑制。还参见图16至图18,关于T1NEO不非特异性地抑制qPCR的附加实验证据,。
图9:构建不能被具有3′->5′核酸外切酶活性的DNA聚合酶延伸的寡核苷酸的困难的说明。通常地,在DNA引物的3′端处的与模板上游区域错配的任何核苷酸将被DNA聚合酶去除,并且引物上的匹配的DNA核苷酸将被延伸。T1NEO的终止子发夹阻止DNA聚合酶识别和加工性地切割T1NEO的3′核苷酸。
图10:对通过具有3′->5′核酸外切酶活性的DNA聚合酶的酶促延伸不太有效地或无效地抑制的寡核苷酸。第一和第二左上图显示了在3′端具有3-碳间隔子(5′-TAAACACCAAGACGTGGTAAATATTTACCTGG/3SpC3/;SEQ ID NO:251)或4nt-TA尾部序列(5′-GACTATGTGACAAAATAGCTAAGGATACAGGAAATATTTAA-3′;SEQ ID NO:252)的引物。尽管这些引物设计有效地防止了通过基于Taq的DNA聚合酶的延伸,但它不能防止通过具有3′->5′核酸外切酶活性的Phusion DNA聚合酶的延伸。中间和底部的图说明了T1NEO设计(中间:5′-ACTGCTGcAGGCGCCCTGTACACTTTAACTCCGCAAGGA-3′;SEQ ID NO:253;底部:5′-ACTGCTGcAGGCGCCCTGTACACTTTAACTCAATCGCAAGATTGA-3′;SEQ ID NO:254),其具有较长和较短的终止子发夹茎序列,或不同的环序列,其在防止Phusion DNA聚合酶延伸方面不太有效。
图11:包含2型不可延伸的寡核苷酸(T2NEO)的关键试剂组分。虚线框表示T2NEO。DNA模板从5′至3′连续地具有上游序列和探针结合序列。模板序列的左侧和T2NEO右侧的灰色箭头表示寡核苷酸的3′端。从5′至3′,T2NEO包括结合序列、错配序列(MS)和尾部序列。模板上的结合序列和探针结合序列大部分或完全反向互补。MS包括彼此反向互补的第一茎序列和第二茎序列。尾部序列与上游序列的反向互补序列不同源。该系统还包含DNA聚合酶。
图12:包括堆叠的发夹、多重发夹或不在发夹结构中的序列的T2NEO的实施例。
图13:在MS和9nt尾部序列中具有两个发夹的T2NEO的实验证明。此处,使用具有3′->5′核酸外切酶活性的Phusion高保真度DNA聚合酶,并且使用Syto-13嵌入染料,将定量PCR(qPCR)应用于NA18537人类基因组DNA模板。在灰色线中,正向引物(FP;5′-ACCAATGGGAGTCACTGCTG-3′;SEQ ID NO:84)和反向引物(RP;5′-TAAGTGGAAAGAACTGGGGTGTC-3′;SEQ ID NO:164)显示出有效地进行qPCR扩增。相反,当FP被T2NEO(5′-ACTGCTGCAGGCGCCCTGTCTGAGAACATTAGTTCTCAGCCTGAGAACATTAGTTCTCAGTACCCCACT-3′;SEQ ID NO:255)替换时,即使当使用10倍高浓度的T2NEO,也没有发生可观察到的PCR扩增。这些数据支持这样的假设,即T2NEO甚至不太可能被具有3′->5′核酸外切酶活性的DNA聚合酶酶促延伸。T2NEO迹线中缓慢和晚期的荧光增加可能是由于RP引物二聚体或基因组上的非特异性扩增。
图14:具有分支发夹结构的T2NEO的实施例和实验证明。(5′-GACTATGTGAcAAAATAGCTAAGGATACAGGAAATAT ACGCAGG CTGAGAACATTAGTTCTCAG TGACCCTAATTATAGGGTCACCTGCGT TGGCAAGAG-3′;SEQ ID NO:256)。
图15:通过阻断剂置换扩增(BDA)将NEO用作变体等位基因富集的阻断剂。在BDA中,不可延伸的阻断剂寡核苷酸在序列上与正向引物重叠,因此阻断剂和正向引物在结合至DNA模板方面竞争。阻断剂被设计成优先地结合至背景DNA模板(野生型),而不太有利地结合至靶DNA模板(变体)。因此,正向引物将优先地扩增靶DNA模板,允许来自靶DNA模板的扩增子比来自背景DNA模板的扩增子富集。如果阻断剂被酶促延伸,那么很大一部分扩增子将对应于阻断剂延伸产物,降低了BDA富集的有效性。
图16:在BDA中使用T1NEO作为阻断剂的BDA的实验证明。此处,NA18537人类基因组DNA用作背景DNA模板,并且NA18562人类基因组DNA用作靶DNA模板。T1NEO(5′-ACTGCTGCAGGCGCCCTGT CGTAAGTCAT TGA GAGAACATTAGTTCTC CT TATA GCAA GAGA-3′;SEQID NO:257)涵盖了rs10230708单核苷酸多态性(SNP)基因座,其中NA18537对于对应与T1NEO上的C核苷酸的模板序列上G等位基因是纯合的,并且NA18562对于与T1NEO错配的T等位基因是纯合的。在没有T1NEO的情况下,NA18537和NA18562两者都有效地扩增,循环阈值(Ct)值为约23.3。当存在T1NEO时,NA18562 gDNA仍然有效地扩增,Ct为24.3,但是NA18537gDNA被抑制扩增,Ct值为38.1。
图17:在BDA中使用T2NEO作为阻断剂的BDA的实验证明。T2NEO(5′-ACTGCTGCAGGCGCCCTGTCTGAGAACATTAGTTCTCAGCCTGAGAACATTAGTTCTCAGTACCCCACT-3′;SEQ ID NO:255)涵盖rs10230708单核苷酸多态性(SNP)基因座,其中NA18537对于G等位基因是纯合的,并且NA18562对于T等位基因是纯合的。
图18:NEO作为用于抑制假基因扩增的方法的实施例。NEO与假基因特异性序列完美匹配,并且正向引物与相应的真基因序列完美匹配。
图19:NEO作为NGS靶富集的杂交捕获探针的实施例。5′-生物素化的NEO探针经由生物素-链霉亲和素相互作用与链霉亲和素包被的磁珠结合,并用于选择性地杂交对应于目的基因的附加衔接子的DNA分子。在杂交捕获富集后的后续珠上PCR扩增中,NEO探针未被延伸。
图20:包含T1NEO的亚型和该亚型的三个实施例的发明的关键组分。在顶部图中,虚线框表示该亚型的结构。DNA模板从5′至3′连续地具有上游序列和探针结合序列。模板序列的左侧和T1NEO亚型的右侧的灰色箭头表示寡核苷酸的3′端。亚型上的生物序列和模板上的探针结合序列大部分或完全反向互补。最3′区域的终止子发夹具有与第二茎序列反向互补的第一茎序列。生物序列和终止子发夹之间的中间发夹具有与第四茎序列反向互补的第三茎序列。中间发夹和终止子发夹分别在第一茎序列和第二茎序列之间具有第一环序列,在第三茎序列和第四茎序列之间具有第二环序列,如终止子发夹和中间发夹的顶部的弧线所示。该系统还包含DNA聚合酶。从第二图到底部的图,显示了三个NEO序列的结构和序列。第二图,MiddleA NEO序列,具有序列5′-GAGAACATTAGTTCTC GTTAGCAATAAC-3′(SEQ IDNO:258)。第三图,MiddleB NEO序列,具有序列5′-GAGAACATTAGTTCTC GATTGCAAAATC-3′(SEQ ID NO:259)。底部图,MiddleC NEO序列,具有序列5′-CCTGTACACATACAGGGTTAGCAATAAC-3′(SEQ ID NO:260)。
图21:MiddleA、MiddleB和MiddleC NEO序列的实验证明。此处,使用具有3′->5′核酸外切酶活性的Phusion高保真度DNA聚合酶,并且使用Syto-13嵌入染料,将定量PCR(qPCR)应用于NA18537人类基因组DNA模板。在灰色线中,显示正向引物(FP)和反向引物(RP)有效地进行qPCR扩增。相反,当FP被MiddleA(SEQ ID NO:81)、MiddleB(SEQ ID NO:2)或MiddleC(SEQ ID NO:82)NEO序列中的任一者替换时,即使当使用10倍高浓度的NEO序列,也不会发生可观察到的PCR扩增。这些数据支持这样的假设,即这些NEO序列甚至都不可能被具有3′->5′核酸外切酶活性的DNA聚合酶酶促延伸。
图22:NEO序列在BDA qPCR和BDANGS的应用。如顶部图所示,NA18537人类基因组DNA用作背景DNA模板,并且NA18562人类基因组DNA用作靶DNA模板。MiddleC NEO序列(SEQID NO:83)覆盖rs10230708单核苷酸多态性(SNP)基因座,其中NA18537对于对应于MiddleCNEO序列上C核苷酸的模板序列上的G等位基因是纯合的,并且NA18562对于与MiddleC NEO序列错配的T等位基因是纯合的。当存在MiddleC NEO序列时,NA18562 gDNA被有效地扩增,Ct为23.3,但是NA18537 gDNA被抑制扩增,Ct值为33.4。如底部图所示,总结了使用80-plexPCR靶富集的实验NGS结果。此处,针对人类基因组的80个不同区域设计了80个不同的正向引物(表2)和80个不同的反向引物(表3)。然后设计80种具有不同生物序列的MiddleB NEO序列阻断剂(表1),以富集变体扩增子。对于同样的0.7%VAF样品,几乎200倍以上的变体将被MiddleB NEO序列富集。
具体实施方式
本文公开了在其3′端处或附近具有合理设计的二级结构的不可延伸的寡核苷酸(NEO)。这些NEO没有3′化学修饰,并且未通过具有3′->5′核酸外切酶活性的高保真度DNA聚合酶进行酶促延伸。这些NEO可以与许多现有方法组合,实现较低的成本。例如,NEO可以与阻断剂置换扩增(BDA)技术一起用于qPCR、下一代测序和纳米孔测序。作为另一实例,NEO可以用于NGS靶富集的杂交捕获探针组。
I.定义
如本文所用,“扩增”是指用于增加一个或多个核苷酸序列拷贝数的任何体外过程。核酸扩增导致核苷酸掺入到DNA或RNA中。如本文所用,一个扩增反应可以由许多轮DNA复制构成。例如,一个PCR反应可以由30-100个变性和复制的“循环”构成。
“聚合酶链式反应”或“PCR”意指通过DNA互补链的同时引物延伸来体外扩增特异性DNA序列的反应。换言之,PCR是用于产生引物结合位点侧接的靶核酸的多个拷贝或复制的反应,此类反应包括以下步骤的一次或多次重复:(i)使该靶核酸变性,(ii)使引物退火至引物结合位点,以及(iii)在核苷三磷酸存在的情况下,通过核酸聚合酶延伸该引物。通常,将反应在热循环仪器中在针对每个步骤优化的不同温度下进行循环。具体的温度、每个步骤的持续时间以及步骤之间的变化速率取决于本领域普通技术人员所熟知的许多因素,例如,参考文献所例示的:McPherson等人编辑,PCR:实用方法和PCR2:(牛津大学的IRL出版社(IRL Press,Oxford),分别出版于1991年和1995年)。
“引物”意指天然的或合成的寡核苷酸,该天然的或合成的寡核苷酸在与多核苷酸模板形成双链体时能够充当核酸合成的起始点,并从其3′端沿着模板延伸,从而形成延伸的双链体。延伸过程期间所添加的核苷酸的序列由模板多核苷酸的序列确定。通常引物通过DNA聚合酶延伸。引物的长度通常与其在引物延伸产物合成中的用途相容,并且长度通常介于8至100个核苷酸之间的范围内,如10至75个、15至60个、15至40个、18至30个、20至40个、21至50个、22至45个、25至40个等,更通常地长度介于18-40个、20-35个、21-30个核苷酸之间的范围内,以及所叙述范围之间的任何长度。通常引物的长度可以在介于10-50个核苷酸之间的范围内,例如15-45个、18-40个、20-30个、21-25个等,以及所叙述范围之间的任何长度。在一些实施例中,引物的长度通常不超过约10个、12个、15个、20个、21个、22个、23个、24个、25个、26个、27个、28个、29个、30个、35个、40个、45个、50个、55个、60个、65个或70个核苷酸。
如本文所用,“掺入”意指成为核酸聚合物的一部分。
如本文所用,术语“在不存在外源性操纵的情况下”是指在不改变核酸分子被修饰的溶液的情况下对核酸分子进行修饰。在具体实施例中,其在没有人手或者在不存在改变溶液条件的机器的情况下发生,该溶液条件也可以被称为缓冲条件。然而,在修饰期间,温度可能会发生变化。
“核苷”是碱基-糖组合,即缺少磷酸的核苷酸。本领域公认术语核苷和核苷酸的用法有一定的互换性。例如,核苷酸脱氧尿苷三磷酸,dUTP,是脱氧核苷三磷酸。掺入到DNA中后,其充当DNA单体,形式上是脱氧尿苷酸,即dUMP或脱氧尿苷酸。可以说,即使在所得DNA中不存在dUTP部分,也可以将dUTP掺入到DNA中。类似地,可以说,即使其仅是底物分子的一部分,也可以将脱氧尿苷掺入到DNA中。
如本文所用,“核苷酸”是指碱基-糖-磷酸盐组合的专业术语。核苷酸是核酸聚合物,即,DNA和RNA的单体单元。该术语包含核苷三磷酸,如rATP、rCTP、rGTP或rUTP,以及脱氧核苷三磷酸,如dATP、dCTP、dUTP、dGTP或dTTP。
术语“核酸”或“多核苷酸”通常是指包括至少一个核碱基的DNA、RNA、DNA-RNA嵌合体或其衍生物或类似物的至少一个分子或链,例如在DNA(例如,腺嘌呤“A”、鸟嘌呤“G”、胸腺嘧啶“T”和胞嘧啶“C”)或RNA(例如,A、G、尿嘧啶“U”和C)中发现的天然存在的嘌呤或嘧啶碱基。术语“核酸”涵盖术语“寡核苷酸”和“多核苷酸”。如本文所用,“寡核苷酸”统称并可互换地指本领域的两个术语,“寡核苷酸”和“多核苷酸”。应当注意,尽管寡核苷酸和多核苷酸是本领域的不同术语,但是其之间没有确切的分界线,并且其在本文中可以互换地使用。术语“衔接子”也可以与术语“寡核苷酸”和“多核苷酸”互换地使用。另外,术语“衔接子”可以指示线性衔接子(单链或双链)或茎环衔接子。这些定义通常是指至少一个单链分子,但是在具体实施例中,这些定义还将涵盖与至少一个单链分子部分、基本上或完全互补的至少一条另外的链。因此,核酸可以涵盖包括有包括分子链的特定序列的一个或多个互补链或“互补序列”的至少一个双链分子或至少一个三链分子。如本文所用,单链核酸可以用前缀“ss”表示,双链核酸用前缀“ds”表示,并且三链核酸用前缀“ts”表示。
“核酸分子”或“核酸靶分子”是指包含标准典范碱基、高度修饰碱基、非天然碱基或它们的碱基的任何组合的任何单链或双链核酸分子。例如但不限于,核酸分子含有四种典范DNA碱基-腺嘌呤、胞嘧啶、鸟嘌呤和胸腺嘧啶,和/或四种典范RNA碱基-腺嘌呤、胞嘧啶、鸟嘌呤和尿嘧啶。当核苷含有2′-脱氧核糖基时,尿嘧啶可以取代胸腺嘧啶。核酸分子可以从RNA转化成DNA,也可以从DNA转化成RNA。例如但不限于,可以使用逆转录酶将mRNA创建成互补DNA(cDNA),并且使用RNA聚合酶将DNA创建成RNA。核酸分子可以是生物来源或合成来源的。核酸分子的实例包含基因组DNA、cDNA、RNA、DNA/RNA杂交体、扩增的DNA、预先存在的核酸文库等。核酸可以从人类样品,如血液、血清、血浆、脑脊液、脸颊刮屑、活检、精液、尿液、粪便、唾液、汗液等中获得。核酸分子可以经受各种处理,如修复处理和片段化处理。片段化处理包含机械剪切、声速剪切和水力剪切。修复处理包含经由延伸和/或连接的缺口修复、抛光以产生平端、去除受损的碱基,如脱氨基、衍生化、脱碱基或交联的核苷酸等。还可以使目的核酸分子经受化学修饰(例如,亚硫酸氢盐转化、甲基化/脱甲基化)、延伸、扩增(例如PCR、等温等)等。
“互补的”或“互补序列”核酸是能够根据标准沃森-克里克(Watson-Crick)、Hoogsteen(胡斯坦碱)或反向胡斯坦碱结合互补规则进行碱基配对的核酸。如本文所用,术语“互补的”或“互补序列”可以指如可以通过上述相同的核苷酸比较进行评估的基本上互补的核酸。术语“基本上互补的”可以指包括至少一个连续核碱基序列的核酸,或者如果分子中不存在一个或多个核碱基部分,则半连续性核碱基能够与至少一条核酸链或双链体杂交,即使并非所有的核碱基均不与对应的核碱基碱基配对。在某些实施例中,“基本上互补的”核酸含有至少一个序列,其中约70%、约71%、约72%、约73%、约74%、约75%、约76%、约77%、约77%、约78%、约79%、约80%、约81%、约82%、约83%、约84%、约85%、约86%、约87%、约88%、约89%、约90%、约91%、约92%、约93%、约94%、约95%、约96%、约97%、约98%、约99%至约100%以及其中任何范围的核碱基序列能够在杂交期间与至少一个单链或双链核酸分子进行碱基配对。在某些实施例中,术语“基本上互补的”是指可以在严格条件下与至少一种核酸链或双链体杂交的至少一种核酸。在某些实施例中,“部分互补的”核酸包括可在低严格条件下与至少一种单链或双链核酸杂交的至少一个序列,或者含有其中少于约70%的核碱基序列的至少一个序列,所述核碱基序列能够在杂交期间与至少一种单链或双链核酸分子进行碱基配对。
术语“非互补的”是指缺乏通过特异性氢键形成至少一个沃森-克里克碱基对的能力的核酸序列。
如本文所用,术语“退化的”是指核苷酸或一系列核苷酸,其中同一性可以选自与所定义的序列相反的多种选择的核苷酸。在具体实施例中,可以从两种或更多种不同的核苷酸中进行选择。在另外的具体实施例中,在一个特定位置处选择核苷酸包括仅从嘌呤、仅从嘧啶、或从非配对的嘌呤和嘧啶中选择。
如本文所用,术语“二级结构”是指碱基对之间的一组相互作用。例如,在DNA双螺旋中,两条DNA链通过氢键连接在一起。二级结构负责核酸呈现的形状。对于单链核酸,最简单的二级结构是线性的。对于线性二级结构,核酸分子中没有两个子序列形成比-2kcal/mol更强的分子内结构。作为单链核酸的另一实例,核酸分子的一部分可以与同一核酸分子的第二部分杂交,从而形成发夹至茎环的二级结构。对于非线性二级结构,核酸分子的至少两个子序列来自比-2kcal/mol更强的分子内结构。
如本文所用,术语“阻断剂寡核苷酸”是指长度为约12至约100个核苷酸的至少一条连续链,并且如果在本文中有所指示,则可以进一步包含防止在扩增过程如聚合酶链式反应期间的酶促延伸的在其3′端处的官能团或核苷酸序列。
如本文所用,术语“引物寡核苷酸”是指这样的分子,该分子包括长度为约12至约100个核苷酸的至少一条连续链,并且足以允许在扩增过程如聚合酶链式反应期间的酶促延伸。
如本文所用,术语“靶中性子序列”是指与靶核酸和背景核酸两者中的序列互补的核苷酸的序列。例如,靶向扩增的所期望的核酸序列(靶核酸)可以存在于具有与靶核酸具有主要同源序列的核酸分子的样品中,除了可变区(背景核酸)外,此类可变区在一些情况下与靶核酸只有单个核苷酸的差异。在此实例中,靶中性子序列与两个核酸之间共享的同源序列的至少一部分互补,但不与可变区互补。因此,如本文所用,术语“阻断剂可变子序列”是指与背景核酸的可变区互补的阻断剂寡核苷酸的核苷酸序列。
如本文所用,术语“重叠子序列”是指引物寡核苷酸的至少5个核苷酸的核苷酸序列,该引物寡核苷酸与如本文所描述的组合物中使用的阻断剂寡核苷酸序列的一部分同源。引物寡核苷酸的重叠子序列可以与阻断剂寡核苷酸的靶中性子序列的任何部分同源,无论是阻断剂可变子序列的5′还是3′。因此,术语“非重叠子序列”是指不是重叠子序列的引物寡核苷酸的序列。
如本文所用,术语“靶序列”是指含待扩增、鉴定或以其他方式分离的所期望的等位基因,如单核苷酸多态性的核酸的核苷酸序列。如本文所用,术语“背景序列”是指不含所期望的等位基因的核酸的核苷酸序列。例如,在一些情况下,背景序列含野生型等位基因,而靶序列含突变等位基因。因此,在一些情况下,背景序列和靶序列源自基因组中的共同基因座,使得除了含在两者之间变化的所期望的等位基因、核苷酸或组或核苷酸的区域之外,每个序列可以是基本上同源的。在另一实例中,在一些情况下,背景序列含假基因序列,而靶序列含真基因序列。
“样品”意指从含有目的核酸的新鲜或保存的生物样品或合成创建源中获得或分离的材料。样品可以包含至少一个细胞、胎儿细胞、细胞培养物、组织样本、血液、血清、血浆、唾液、尿液、泪液、阴道分泌物、汗液、淋巴液、脑脊液、粘膜分泌物、腹膜液、腹水液、粪便物、身体渗出液、脐带血、绒毛膜绒毛、羊水、胚胎组织、多细胞胚胎、裂解物、提取物、溶液或怀疑含有目的免疫核酸的反应混合物。样品还可以包含非人类来源,如非人灵长类动物、啮齿类动物和其他哺乳动物、其他动物、植物、真菌、细菌和病毒。
如本文所用,关于核苷酸序列,“基本上已知的”是指具有足够的序列信息,以便允许制备包含其扩增的核酸分子。这通常为约100%,尽管在一些实施例中,衔接子序列的一些部分是随机的或退化的。因此,在具体实施例中,基本上已知的是指约50%至约100%、约60%至约100%、约70%至约100%、约80%至约100%、约90%至约100%、约95%至约100%、约97%至约100%、约98%至约100%或约99%至约100%。
如本文所用,就指定组分而言,“本质上不含”在本文中用于意指指定组分没有被故意调配成组合物和/或仅作为污染物或以微量存在。因此,由组合物的任何非预期污染产生的特定组分的总量远低于0.05%,优选地低于0.01%。最优选的是其中使用标准分析方法检测不到指定组分的量的组合物。
如本文中说明书中所用,“一个/一种(a)”或“一个/一种(an)”可以意指一个或多个/一种或多种(one or more)。如本文中权利要求中所用,当与单词“包括(comprising)”结合使用时,单词“一个/一种”或“一个/一种”可以意指一个或多于一个。
权利要求中术语“或”的使用用于意指“和/或”,除非明确指出仅指替代方案或替代方案是相互排斥的,但本公开支持仅指替代方案以及“和/或”的定义。如本文所用,“另一个”可以意指至少第二个或更多个。
贯穿本申请,术语“约”是用于指示一个值包含用于测定值的装置、方法的固有的误差变化,或研究受试者间存在的变化或在所陈述的值10%内的值。
II.不可延伸的寡核苷酸(NEO)
A.1型NEO
1型不可延的伸寡核苷酸(T1NEO)具有5′结合序列,其显示出与DNA模板序列的探针结合序列的反向互补序列显著的序列相似性(图1)。T1NEO结合序列可以与DNA模板序列的探针结合序列的反向互补序列70%、75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%相同。T1NEO的最3′区域具有终止子发夹区域,该终止子发夹区域包括位于第一茎序列和第二茎序列之间的环序列。第一茎序列和第二茎序列彼此反向互补。终止子发夹不与DNA模板序列的上游序列反向互补。在一个实施例中,终止子发夹具有序列5′-TCTC GCAA GAGA-3′(SEQ ID NO:244;图2)。
T1NEO的不同实施例在图3至图5中显示。在这些变体实施例中,T1NEO进一步包括位于结合序列和终止子发夹之间的错配序列(MS)。该MS可以包括两个或更多个发夹结构(图5),一个发夹结构(图4),或者没有发夹结构(图3)。此外,图20显示了T1NEO的特定亚型,其包括生物(结合)序列和终止子发夹之间的中间发夹。
高保真度DNA聚合酶的3′->5′核酸外切酶活性是使这些酶能够用于检测和定量具有低变体等位基因频率(VAF)的突变(如肿瘤组织或无细胞DNA中的体细胞突变)的关键特征。3′->5′核酸外切酶活性允许动力学校正,由此可以去除在生长扩增子的3′端不正确地并入的DNA核苷酸,并且使DNA聚合酶如Phusion和Q5表现出比基于Taq的DNA聚合酶低20倍至200倍的错误并入错误率。然而,这种3′->5′核酸外切酶活性也使得设计不旨在被酶促延伸的DNA探针和阻断剂具有挑战性(图9)。甚至许多阻止Taq延伸的3′化学修饰也不能有效阻止3′->5′核酸外切酶活性后的延伸。图10显示了一系列具有和不具有3′化学修饰的DNA寡聚体,该化学修饰在防止通过具有3′->5′核酸外切酶活性的DNA聚合酶的酶促延伸方面不太有效。
然而,T1NEOs不能有效地被DNA聚合酶(包含具有3′->5′核酸外切酶活性的高保真度DNA聚合酶)延伸。使用T1NEO和反向引物(ACATGGTTAGATATTAGCCTGACCTATG;SEQ IDNO:165)进行许多定量PCR(qPCR)实验,以便证明这一点(图6至图8)。没有qPCR扩增或非常晚的扩增表明T1NEO未被酶促延伸。相反,使用与T1NEO序列相似但是缺少终止子发夹的正向引物(GAGGGGTATTAGAAGAATGACTATGTGA SEQ ID NO:85),显示出有效的qPCR扩增和检测,表明引物设计、DNA聚合酶和DNA输入样品都与PCR扩增兼容。此外,使用三个列出的序列(MiddleA、MiddleB、MiddleC NEO序列)及其相应的反向引物进行许多定量PCR(qPCR)实验(图21)。对于包括中间发夹的T1NEO,没有qPCR扩增或非常晚的扩增表明该亚型未被酶促延伸。相反,使用序列与NEO序列相似但是缺少中间发夹和终止子发夹的正向引物,显示出有效的qPCR扩增和检测,表明引物设计、DNA聚合酶和DNA输入样品都与PCR扩增兼容。在一些实施例中,嵌入DNA染料(例如,Syto-13)可以用于检测扩增,该染料对dsDNA扩增子的积累非特异性地产生荧光。在其他实施例中,qPCR反应包括Taqman探针,而不是嵌入染料,以报告特异性扩增子积累。
表1 具有中间发夹的示例性T1NEO。
/>
/>
/>
B.2型NEO
2型不可延伸的寡核苷酸(T2NEO)类似于T1NEO,除了它在3′端不包括终止子发夹。而是,它包括有包括发夹序列的错配序列和不包括发夹的3′尾部序列(图11)。T2NEO的各种实施例在图12和图14中显示图13和图14显示了序列和实验qPCR结果,证明T2NEO未被Phusion DNA聚合酶有效延伸。
III.NEO
A.阻断剂在阻断剂置换扩增(BDA)等位基因富集中的应用
BDA使用不可延伸的阻断剂,该阻断剂具有与预期的野生型模板序列完美匹配的序列。当不可延伸的寡核苷酸与模板结合时,正向引物不能有效地与模板结合,因为与NEO结合的模板序列的一部分也是与正向引物结合的子序列。在一些实施例中,正向引物结合至的模板的子序列具有1个核苷酸至20个核苷酸的少量核苷酸,这些核苷酸不涵盖在NEO结合至的模板的子序列中。
如果模板序列甚至具有单个核苷酸序列变体,则在模板和结合序列中的NEO之间形成的错配气泡引起热力学不稳定,该热力学不稳定导致正向引物比NEO更有利地与模板结合。以T1NEO为例,当在模板上存在由于序列变体而形成TC错配气泡时,T1NEO被正向引物从模板上置换。在一些实施例中,正向引物然后能够被DNA聚合酶延伸。在一些实施例中,野生型模板分子和变体模板分子的混合物存在于模板样品中,并且将BDA和TEO应用于样品导致变体模板通过变体模板的选择性扩增而比野生型模板富集。在一些实施例中,DNA聚合酶是热稳定的DNA聚合酶,并且通过聚合酶链式反应(PCR)实现扩增。
为了证明包括中间发夹亚型的T1NEO可以应用于BDA,包含qPCR和高通量测序,使用亚型NEO序列及其相应的正向引物和反向引物进行定量PCR(qPCR)和下一代测序(NGS)实验(图22)。顶部的图显示了实验qPCR结果。靶DNA模板NA18562人类基因组DNA比背景DNA模板NA18537人类基因组DNA富集。当存在MiddleC NEO序列时,NA18562 gDNA被有效扩增,Ct为23.3,但是NA18537 gDNA被抑制扩增,Ct值为33.4。如底部图中所示,总结了使用80-plexPCR靶富集的实验NGS结果。此处,针对人类基因组的80个不同区域设计了80个不同的正向引物和80个不同的反向引物。然后设计80种具有不同生物序列的MiddleB NEO序列阻断剂来富集变体扩增子。对于同样的0.7%VAF样品,几乎200倍以上的变体将被MiddleB NEO序列富集。
在一些方面,作为BDA阻断剂的NEO可以包括靶向假基因或其他不期望的基因组区域的序列和阻止通过DNA聚合酶的延伸的3′序列或修饰,从而抑制假基因扩增。例如,NEO可以与假基因特异性序列完美匹配,并且正向引物与相应的真基因序列完美匹配(图18)。
对于使用NEO作为阻断剂的BDA,将BDA正向引物、NEO、反向引物、DNA聚合酶、dNTP和PCR缓冲液与模板样品混合用于BDA扩增。然后,在足以实现核酸扩增的条件下进行BDA扩增持续2、3、4、5、6、7、8、9、10、11、12、13、14、15、16、17、18、19、20、21、22、23、24、25、26、27、28、29、30、2-30、2-25、2-23、2-20、2-15、4-30、4-25、4-23、4-20、4-15、6-30、6-25、6-23、6-20、6-15、8-30、8-25、8-23、8-20、8-15、10-30、10-25、10-23、10-20或10-15个循环的BDA扩增。
作为BDA阻断剂,NEO可以包含具有靶中性(即野生型)子序列和阻断剂可变(即靶)子序列的第一序列。在一些方面,可变子序列包含至少一个核苷酸、至少两个核苷酸、至少三个核苷酸、至少四个核苷酸或至少五个核苷酸。然而,在一些情况下,如果待检测的靶核酸用于检测插入,则NEO可能不包含阻断剂可变子序列。NEO可变子序列在其3′端和5′端侧接靶中性子序列,并且与靶中性子序列是连续的。
BDA正向引物足以诱导未被NEO结合的模板核酸的酶促延伸。BDA正向引物的3′端包含与NEO的5′端重叠的序列。NEO与BDA正向引物的3′端重叠的部分仅由靶中性子序列组成,因此BDA正向引物可以不包含任何与阻断剂可变子序列同源的序列。
BDA正向引物和NEO的序列可以基于其与靶核酸序列(例如,期望其扩增或检测的序列,例如SNP、插入、缺失或任何其他突变)和变体核酸序列(例如,寻求抑制其扩增的序列,例如野生型序列)杂交的热力学进行合理设计。在一些实施例中,NEO以明显高于BDA正向引物的浓度存在,因此在与引物结合之前,绝大多数的靶和变体核酸序列与NEO结合。BDA正向引物与BDA阻断剂-靶分子或BDA阻断剂-变体分子瞬时结合,并且具有在与靶或变体结合时取代NEO的可能性。因为NEO序列对变体靶是特异性的,所以其从变体的置换不如其从野生型的置换在热力学上有利。因此,非等位基因特异性BDA正向引物以比扩增野生型序列更高的产率/效率扩增靶序列。WO2015/179339提供了在设计BDA引物和NEO对时可以考虑的示例性热力学考虑,该文献通过引用整体并入本文。
在一些方面,可以设计NEO和BDA正向引物,使得每种寡核苷酸的结合满足杂交条件的某些标准自由能。例如,BDA正向引物与模板核酸杂交的标准自由能(ΔG°
+2kcal/mol≥ΔG°
在一些方面,可以设计NEO和BDA正向引物,使得ΔG°
在一些方面,可以设计BDA正向引物,使得与NEO结合位点杂交的引物部分具有这样的标准杂交自由能(ΔG°
用于从序列中计算ΔG°值的方法是本领域已知的。计算不同区相互作用的ΔG°存在不同的惯例。WO2015/179339提供了基于最近邻模型的示例性能量计算,该文献通过引用整体并入本文。从引物序列、阻断剂序列、靶序列、变体序列、操作温度和操作缓冲液条件计算ΔG°
在一些方面,BDA正向引物和NEO的长度可以各自独立地为约12-100、约12-90、约12-80、约12-70、约12-60、约12-50、约12-40、约12-30、约15-100、约15-90、约15-80、约15-70、约15-60、约15-50、约15-40、约15-30、约20-100、约20-90、约20-80、约20-70、约20-60、约20-50、约20-40或约20-30个核苷酸。在一些方面,BDA正向引物和NEO的长度可以各自独立地为20、21、22、23、24、25、26、27、28、29、30、31、32、33、34、35、36、37、38、39、40、41、42、43、44、45、46、47、48、49、50、51、52、53、54、55、56、57、58、59或60个核苷酸。
在一些方面,与NEO结合位点杂交的BDA正向引物的部分为约5-40个核苷酸、约7-40、约9-40、约11-40、约13-40、约15-40、约20-40、约25-40、约30-40、约35-40、约5-35、约7-35、约9-35、约11-35、约13-35、约15-35、约20-35、约25-35、约30-35、约5-30、约7-30、约9-30、约11-30、约13-30、约15-30、约20-30、约25-30、约5-25、约7-25、约9-25、约11-25、约13-25、约15-25、约20-25、约5-20、约7-20、约9-20、约11-20、约13-20或约15-20个核苷酸。在一些方面,与NEO结合位点杂交的BDA正向引物的部分为约5、6、7、8、9、10、11、12、13、14、15、16、17、18、19、20、21、22、23、24、25、26、27、28、29、30、31、32、33、34、35、36、37、38、39或40个核苷酸。
在一些方面,NEO的浓度比BDA正向引物的浓度高约2-10000、约2-9000、约2-8000、约2-7000、约2-6000、约2-5000、约2-4000、约2-3000、约2-2000、约2-1000、约2-900、约2-800、约2-700、约2-600、约2-500、约2-400、约2-300、约2-200、约2-150、约2-100、约2-90、约2-80、约2-70、约2-60、约2-50、约2-40、约2-30、约2-20、约2-10、约4-10000、约4-9000、约4-8000、约4-7000、约4-6000、约4-5000、约4-4000、约4-3000、约4-2000、约4-1000、约4-900、约4-800、约4-700、约4-600、约4-500、约4-400、约4-300、约4-200、约4-150、约4-100、约4-90、约4-80、约4-70、约4-60、约4-50、约4-40、约4-30、约4-20、约4-10、约6-10000、约6-9000、约6-8000、约6-7000、约6-6000、约6-5000、约6-4000、约6-3000、约6-2000、约6-1000、约6-900、约6-800、约6-700、约6-600、约6-500、约6-400、约6-300、约6-200、约6-150、约6-100、约6-90、约6-80、约6-70、约6-60、约6-50、约6-40、约6-30、约6-20、约6-10、约10-10000、约10-9000、约10-8000、约10-7000、约10-6000、约10-5000、约10-4000、约10-3000、约10-2000、约10-1000、约10-900、约10-800、约10-700、约10-600、约10-500、约10-400、约10-300、约10-200、约10-150、约10-100、约10-90、约10-80、约10-70、约10-60、约10-50、约10-40、约10-30、约10-20、约20-10000、约20-9000、约20-8000、约20-7000、约20-6000、约20-5000、约20-4000、约20-3000、约20-2000、约20-1000、约20-900、约20-800、约20-700、约20-600、约20-500、约20-400、约20-300、约20-200、约20-150、约20-100、约20-90、约20-80、约20-70、约20-60、约20-50、约20-40、约20-30、约40-10000、约40-9000、约40-8000、约40-7000、约40-6000、约40-5000、约40-4000、约40-3000、约40-2000、约40-1000、约40-900、约40-800、约40-700、约40-600、约40-500、约40-400、约40-300、约40-200、约40-150、约40-100、约40-90、约40-80、约40-70、约40-60或约40-50。
为了多重BDA(mBDA)同时富集多组遗传基因座的潜在靶序列,对于每个基因座采用不同的BDA正向引物和NEO。这些均在溶液中与样品、DNA聚合酶、dNTP和适于PCR的缓冲液同时组合。为了防止基于DNA的PCR抑制,所有寡聚体的总浓度可以保持在50微摩尔以下。PCR反应的退火/延伸步骤的长度与最低BDA正向引物物质的浓度成反比。为了防止方案过长,建议所有BDA正向引物浓度为至少100皮摩尔。每种NEO物质的浓度应该是其相应的BDA正向引物物质的至少2倍。
除了上述单重BDA的标准设计原则之外,多重BDA(mBDA)的寡聚体设计需要进一步考虑,以防止不期望的“引物二聚体”物质。当预测候选序列集表现出非选择性结合相互作用时,mBDA序列设计算法应该惩罚该候选序列集。参见例如WO 2019/164885,该文献通过引用整体并入本文。
表2 示例性的正向引物。
/>
表3.示例性的反向引物。
/>
B.杂交捕获探针
在另一实施例中,NEO可以用作杂交捕获探针,用于NGS应用中的靶富集(图19)。杂交捕获探针包括能够与靶核酸中的独特区域杂交并被捕获到固相上的核酸序列。用NEO这样做允许在杂交捕获富集后进行珠上PCR扩增,而无需考虑探针延伸的混淆影响,因为NEO探针是不可延伸的。为此,NEO可以用5′-生物素化进行功能化,以允许与链霉亲和素包被的磁珠结合。这些NEO提供了一种选择性杂交和捕获对应于目的基因的DNA分子的手段。
杂交捕获探针的各种用途是本领域技术人员所熟知的,并且可以应用于检测和区分各种突变,包含但不限于插入、缺失、倒位、重复序列以及多核苷酸多态性和单核苷酸多态性(SNP)。在一些实施例中,靶向各种序列的一组NEO可以用作杂交捕获探针。例如,一组NEO可以被设计成基于SNP特征来区分不同的人类基因组。在一些实施例中,测试样品可以含有核酸的复杂混合物,其中靶核酸可以对应于在总人类基因组DNA或RNA中含有的目的基因,或者是作为临床样品的次要组分的致病生物体的核酸序列的一部分。
IV.靶核酸的进一步加工
A.DNA的扩增
许多模板依赖性过程可用于扩增存在于给定模板样品中的核酸。最熟知的扩增方法之一是聚合酶链式反应(被称为PCR
B.DNA的测序
还提供了用于衔接子连接片段的文库的测序方法。本领域技术人员已知的任何核酸测序技术都可以用于本公开的方法中。DNA测序技术包含:使用标记的终止子或引物和平板或毛细管中的凝胶分离的经典双脱氧测序反应(桑格法(Sanger method));使用可逆终止的标记核苷酸的边合成边测序;焦磷酸测序;454测序;与标记的寡核苷酸探针文库的等位基因特异性杂交;使用与标记的克隆文库的等位基因特异性杂交以及随后在聚合步骤期间标记的核苷酸的连接和掺入的实时监测的边合成边测序;以及SOLiD测序。
核酸文库可以用与因美纳(Illumina)测序兼容的方法产生,如Nextera
在具体方面,在本公开的方法中使用的测序技术包含来自因美纳有限责任公司的HiSeq
可用于本公开的方法中的DNA测序技术的另一个实例是454测序(罗氏公司(Roche))(Margulies等人,2005)。454测序涉及两个步骤。在第一步骤中,DNA被剪切成大约300-800个碱基对的片段,并且这些片段被平端化。然后将寡核苷酸衔接子与片段的端部连接。衔接子作为引物用于片段的扩增和测序。可以使用例如含有5′-生物素标签的衔接子B将片段与DNA捕获珠,例如链霉亲和素涂覆的珠连接。连接在珠上的片段在油-水乳液的液滴内进行PCR扩增。结果是在每个珠上克隆扩增DNA片段的多个拷贝。在第二步骤中,这些珠被捕获在孔中(皮升大小)。对每个DNA片段平行地进行焦磷酸测序。一个或多个核苷酸的添加产生被测序仪器中的CCD相机记录的光信号。信号强度与掺入的核苷酸数量成正比。
可用于本公开的方法中的DNA测序技术的另一个实例是SOLiD技术(生命技术有限责任公司(Life Technologies,Inc.))。在SOLiD测序中,基因组DNA被剪切成片段,并且衔接子与片段的5′端和3′端连接以产生片段文库。可替代地,可以通过将衔接子与片段的5′端和3′端连接,环化片段,消化经环化的片段以产生内部衔接子,并将衔接子与所得片段的5′端和3′端连接以产生配对文库来引入内部衔接子。接下来,在含有珠、引物、模板和PCR组分的微型反应器中制备克隆珠群体。PCR后,将模板变性并且富集珠以分离具有延伸模板的珠。对选定珠上的模板进行3′修饰,以允许与载玻片结合。
可用于本公开的方法中的DNA测序技术的另一个实例是IonTorrent系统(生命技术有限责任公司)。Ion Torrent使用高密度的微加工孔阵列以大规模并行的方式执行此生化过程。每个孔均有不同的DNA模板。孔下方是离子敏感层,并且下方是专有的离子传感器。如果核苷酸,例如C,被添加到DNA模板中,并且然后被掺入到DNA链中,就会释放出氢离子。来自离子的电荷将会改变溶液的pH,这可以通过专有的离子传感器检测到。测序仪将判定碱基,直接从化学信息转化为数字信息。离子个人基因组机器(PGM
可用于本公开的方法中的测序技术的另一个实例包含太平洋生物科学公司(Pacific Biosciences)的单分子实时(SMRT
另外的测序平台包含CGA平台(全基因组学)。CGA技术是基于环形DNA文库的制备和滚环扩增(RCA)来产生排列在固体载体上的DNA纳米球(Drmanac等人,2009)。全基因组学的CGA平台使用称为组合探针锚定连接(cPAL)的新策略以进行测序。该过程开始于锚分子与独特衔接子之一之间的杂交。四个退化的9聚体寡核苷酸用对应于探针的第一位置中的特异性核苷酸(A、C、G或T)的特异性荧光团标记。序列测定发生在反应中,其中正确匹配的探针与模板杂交,并且使用T4 DNA连接酶与锚连接。连接产物成像后,连接的锚-探针分子变性。使用在n+1、n+2、n+3和n+4位置处含有已知碱基的新的荧光标记的9聚体探针组,将杂交、连接、成像和变性的过程重复五次。
V.试剂盒
本文所描述的技术包含包括如本文公开的不可延伸的寡核苷酸的试剂盒。示例性的试剂盒包含qPCR试剂盒、桑格试剂盒、NGS板和纳米孔测序板。“试剂盒”是指物理元素的组合。例如,试剂盒可以包含,例如,一种或多种组分,如核酸引物、核酸阻断剂、酶、反应缓冲液、说明书和用于实施本文所描述的技术的其他要素。这些物理要素可以以适于实施本发明的任何方式布置。
试剂盒的组分可以以水性介质或冻干形式包装。试剂盒的容器装置将通常包含组分可以放置有于其中并且优选地是适当等分(例如,等分到微量滴定板的孔中)的至少一个小瓶、试管、烧瓶、瓶子、注射器或其他容器装置。当试剂盒中存在多于一种组分时,试剂盒通常还将含有第二、第三或其他另外的容器,另外的组分可以单独放置于其中。然而,单个小瓶中可以包括组分的各种组合。本发明的试剂盒通常还包含一种用于以紧密封闭的方式容纳核酸和任何其他试剂容器以供商业销售的装置。此类容器可以包含将期望的小瓶保留在其中的注射或吹模成型的塑料容器。试剂盒还将包含试剂盒组分的使用说明以及试剂盒中未包含的任何其他试剂的使用说明。说明书可以包含可以实施的变体。
VI.实例
包含了以下实例以证明本发明的优选实施例。本领域的技术人员应当理解,以下实施例中所公开的技术表示由本发明人发现的在本发明的实践中很好地起作用并且因此可以被认为构成本发明的优选实践模式的技术。然而,根据本公开内容,本领域的技术人员应当理解,在不脱离本发明的精神和范围的情况下,可以对所公开的具体实施例进行许多改变并且仍然获得相同或类似的结果。
实例1-定量PCR(qPCR)反应方案和条件
对于此处呈现的实验结果,如前所述,在10μL的反应混合物中,正向引物和反向引物的最终浓度各自为100nM或400nM。如图所示,NEO的最终浓度为100nM、1000nM或4000nM。除非另有说明,否则Phusion Hi-Fi DNA聚合酶、syto 13相互作用染料和Phusion所需的试剂用于所有qPCR实验(新英格兰生物实验室有限责任公司(New England Biolabs,Inc))。输入DNA通常是5ng的NA18537或NA18562人类基因组DNA(Coriell细胞库)。使用Bio-RadCFX96 qPCR仪器进行热循环和荧光测量。热循环方案如下:1.98℃30秒;2.50个循环(98℃持续10秒,60℃持续30秒,72℃持续30秒)。
相同的方案和条件用于使用NEO作为阻断剂的阻断剂置换扩增(BDA)qPCR实验。
我们使用PowerUp SYBR DNA聚合酶Mastermix(Thermo Fisher)进行所有基于Taq的实验。输入DNA通常是5ng的NA18537人类基因组DNA(Coriell细胞库)。使用Bio-RadCFX96 qPCR仪器进行热循环和荧光测量。热循环方案如下:1.95℃3分钟;2.50个循环(95℃持续10秒,60℃持续30秒)。
实例2-下一代测序(NGS)反应方案和条件
图22中总结的NGS实验的数据是使用因美纳MiSeq仪器和MiSeq v2配端150+150循环试剂盒收集的。每个文库使用在50uL反应混合物中的25ng的输入DNA。库准备过程简要总结如下:
1a.对于没有阻断剂的文库,使用Phusion 2X MasterMix(新英格兰生物实验室有限责任公司),使用每丛15nM引物,进行13个循环的BDA PCR(98℃持续10秒,60℃持续5分钟,72℃持续2分钟)。
1b.对于含有阻断剂的文库,使用Phusion 2X MasterMix(新英格兰生物实验室有限责任公司)使用每丛15nM引物和每丛150nM阻断剂,对BDA PCR进行23个循环(98℃持续10秒,60℃持续5分钟,72℃持续2分钟)。
2.使用1.8x SPRI珠进行DNA纯化。
3.使用Phusion 2X MasterMix(新英格兰生物实验室有限责任公司),使用每丛15nM引物,进行2个循环的衔接子PCR(98℃持续10秒,60℃持续5分钟,72℃持续2分钟)。
4.使用1.6x SPRI珠进行DNA纯化。
5a.对于没有阻断剂的文库,使用Phusion 2X MasterMix(新英格兰生物实验室有限责任公司),使用500nM索引引物,进行15个循环的索引PCR(98℃持续10秒,60℃持续60秒,72℃持续60秒)。
5b.对于含有阻断剂的文库,使用500nM索引引物进行12个循环的索引PCR(98℃持续10秒,60℃持续60秒,72℃持续60秒)Phusion 2X MasterMix(新英格兰生物实验室有限责任公司)。
6a.对于没有阻断剂的文库,使用0.6x+0.3x SPRI珠进行DNA纯化。
6b.对于含有阻断剂的文库,使用0.7x+0.3x SPRI珠进行DNA纯化。
实例3-NGS生物信息学分析方法
用于从NGS FASTQ文件中分析NGS读取的方法总结如下:
1.修剪来自每次读取的衔接子序列。
2.计算与野生型扩增子SNP的基因座之前和之后的10nts(WT读段)或变体扩增子的SNP基因座之前和之后的10nts(Var读段)完美匹配的插入读段的数量。读段中的任何退化核苷酸,如N,都被认为是错配的,并且对WT读段或Var读段没有贡献。
3.计算与中间发夹序列、终止子发夹序列或中间发夹序列和终止子发夹序列的完整序列完美匹配的插入读段的数量。总数将计为NEO读段。
实例4-倍数富集分析和VAF定量
变体模板的倍数富集(EF)被定义为变体模板相对于相应野生型模板的相对扩增。一般来说,在ACE的情况下较多次数的PCR循环导致较大的EF值。在NGS文库设置中,VRF、EF和变体等位基因频率(VAF)的值满足以下等式:
VRF=(VAF*EF)/(VAF*EF+(1-VAF))
VAF=(VRF)/(VRF*(1-EF)+EF)
EF=(VRF*(VAF-1))/(VAF*(VRF-1))
给定三个变量中任意两个的已知值,可以计算出最后一个变量。因此,在初始校准实验期间,来自已知样品的VRF和VAF可以用于计算EF。之后,当在具有未知VAF的样品上运行NGS时,VRF和EF可以用来计算VAF的值。
实例5-T1NEO和T2NEO不能被高保真度DNA聚合酶有效延伸
高保真度DNA聚合酶的3′->5′核酸外切酶活性是使这些酶能够用于检测和定量具有低变体等位基因频率(VAF)的突变(如肿瘤组织或无细胞DNA中的体细胞突变)的关键特征。3′->5′核酸外切酶活性允许动力学校正,由此可以去除在生长扩增子的3′端的不正确地并入的DNA核苷酸,并且使DNA聚合酶如Phusion和Q5表现出比基于Taq的DNA聚合酶低20倍至200倍的错误并入错误率。然而,这种3′->5′核酸外切酶活性也使得设计不旨在被酶促延伸的DNA探针和阻断剂具有挑战性(图9)。甚至许多阻止Taq延伸的3′化学修饰也不能有效阻止3′->5′核酸外切酶活性后的延伸。图10显示了一系列具有和不具有3′化学修饰的DNA寡聚体,该化学修饰在防止通过具有3′->5′核酸外切酶活性的DNA聚合酶的酶促延伸方面不太有效。
为了证明T1NEO不能有效地被DNA聚合酶延伸,包含不能被具有3′->5′核酸外切酶活性的高保真度DNA聚合酶延伸,使用T1NEO和反向引物(5′-ACATGGTTAGATATTAGCCTGACCTATG-3′;SEQ ID NO:165)进行了许多定量PCR(qPCR)实验(图6至图8)。没有qPCR扩增或非常晚的扩增表明T1NEO未被酶促延伸。相反,使用与T1NEO序列相似但是缺少终止子发夹的正向引物(5′-GAGGGGTATTAGAAGAATGACTATGTGA-3′;SEQ ID NO:85),显示出有效的qPCR扩增和检测,表明引物设计、DNA聚合酶和DNA输入样品都与PCR扩增兼容。在这些实验中,使用Syto-13嵌入DNA染料,该染料对dsDNA扩增子的积累非特异性地产生荧光。
为了证明具有中间发夹的T1NEO不能有效地被DNA聚合酶延伸,包括不能被具有3′->5′核酸外切酶活性的高保真度DNA聚合酶延伸,使用三个列出的序列(MiddleA(SEQID NO:81)、MiddleB(SEQ ID NO:2)或MiddleC(SEQ ID NO:82)NEO序列)及其相应的反向引物((分别为SEQ ID NOs:204、165和176;图21)进行了许多定量PCR(qPCR)实验。没有qPCR扩增或非常晚的扩增表明该亚型不是酶促延伸的。相反,使用序列与NEO序列相似但是缺少中间发夹和终止子发夹的正向引物,显示出有效的qPCR扩增和检测,表明引物设计、DNA聚合酶和DNA输入样品都与PCR扩增兼容。在这些实验中,使用Syto-13嵌入DNA染料,该染料对dsDNA扩增子的积累非特异性地产生荧光。
为了证明在MS中具有两个发夹和9nt尾部序列的T2NEO(图13)以及具有分支发夹结构的T2NEO(图14)不能被高保真度DNA聚合酶有效延伸,使用具有3′->5′核酸外切酶活性的Phusion高保真度DNA聚合酶并且使用Syto-13嵌入染料,将定量PCR(qPCR)应用于NA18537人类基因组DNA模板。即使当使用10倍高浓度的T2NEO,也没有发生可观察到的PCR扩增(图13和图14)。相反,正向引物和反向引物能够有效地进行qPCR扩增(图13和图14)。因此,T2NEO甚至不能被具有3′->5′核酸外切酶活性的DNA聚合酶酶促延伸。T2NEO迹线中缓慢和晚期的荧光增加可能是由于RP引物二聚体或基因组上的非特异性扩增。
实例6-作为BDA阻断剂的NEO
BDA使用不可延伸的阻断剂,该阻断剂具有与预期的野生型模板序列完美匹配的序列。在BDA中,该不可延伸的阻断剂寡核苷酸在序列上与正向引物重叠,因此阻断剂和正向引物在结合至DNA模板方面竞争。当不可延伸的寡核苷酸与模板结合时,正向引物不能有效地与模板结合,因为与NEO结合的模板序列的一部分也是与正向引物结合的子序列。在一些实施例中,正向引物结合至的模板的子序列具有少量核苷酸,在1个核苷酸和20个核苷酸之间,不涵盖在NEO结合至的模板的子序列内。
如果模板序列甚至具有单个核苷酸序列变体,则在模板和结合序列中的NEO之间形成的错配气泡导致热力学不稳定,这导致正向引物与模板的结合比NEO与模板的结合更有利(图15)。以T1NEO为例,当在模板上存在由于序列变体形成的TC错配气泡时,T1NEO被正向引物从模板上置换。在一些实施例中,正向引物然后能够被DNA聚合酶延伸。在一些实施例中,野生型模板和变体模板分子的混合物存在于模板样品中,并且将BDA与NEO一起应用于样品导致变体模板通过变体模板的选择性扩增而比野生型模板富集(图15)。在一些实施例中,DNA聚合酶是热稳定的DNA聚合酶,并且通过聚合酶链式反应(PCR)实现扩增。
为了使用T1NEO证明这一点,将NA19537人类基因组DNA用作野生型模板,并且将NA18562人类基因组DNA用作变体模板。T1NEO被设计为覆盖rs10230708单核苷酸多态性(SNP)基因座,其中NA18537对于对应于T1NEO上C核苷酸的模板序列上的G等位基因是纯合的,并且NA18562对于T等位基因是纯合的,这与T1NEO是错配的。在不存在T1NEO的情况下,NA18537和NA18562均有效扩增,循环阈值(Ct)值为约23.3(图16)。当存在T1NEO时,NA18562gDNA仍然有效扩增,Ct为24.3,但是NA18537 gDNA被抑制扩增,Ct值为38.1(图16)。
为了证明具有中间发夹的T1NEO可以应用于BDA,包含qPCR和高通量测序,使用该亚型NEO序列及其相应的正向引物和反向引物进行定量PCR(qPCR)和下一代测序(NGS)实验(图22)。顶部的图显示实验qPCR结果(使用根据SEQ ID NO:83的NEO;根据SEQ ID NO:84的正向引物和根据SEQ ID NO:164的反向引物。靶DNA模板NA18562人类基因组DNA比背景DNA模板NA18537人类基因组DNA富集。当存在MiddleC NEO序列时,NA18562 gDNA被有效扩增,Ct为23.3,但是NA18537 gDNA被抑制扩增,Ct值为33.4。如底部图中所示,总结了使用80-plex PCR靶富集的实验NGS结果。此处,针对人类基因组的80个不同区域设计了80个不同的正向引物和80个不同的反向引物。然后设计80种具有不同生物序列的MiddleB NEO序列阻断剂来富集变体扩增子。对于同样的0.7%VAF样品,几乎200倍以上的变体将被MiddleBNEO序列富集。
为了使用T2NEO证明这一点,再次使用NA19537人类基因组DNA作为野生型模板,并且再次使用NA18562人类基因组DNA作为变体模板。T2NEO被设计为覆盖rs10230708单核苷酸多态性(SNP)基因座,其中NA18537对于对应于T2NEO上C核苷酸的模板序列上的G等位基因是纯合的,并且NA18562对于T等位基因是纯合的,这与T2NEO是错配的。在不存在T2NEO的情况下,NA18537和NA18562两者都被有效扩增,循环阈值(Ct)值为约23.2(图17)。当存在T2NEO时,NA18562 gDNA仍然被有效扩增,Ct为27.1,但是NA18537 gDNA被抑制扩增,其中Ct值为40.4(图17)。
在一些方面,作为BDA阻断剂的NEO可以包括靶向假基因或其他不期望的基因组区域的序列和阻止通过DNA聚合酶的延伸的3′序列或修饰,从而抑制假基因扩增。例如,NEO可以与假基因特异性序列完美匹配,并且正向引物与相应的真基因序列完美匹配(图18)。
* * *
根据本公开,无需过度实验即可制备和执行本文所公开和要求保护的所有方法。虽然已经根据优选实施例描述了本发明的组合物和方法,但是对于本领域的技术人员来说将显而易见的是,在不脱离本发明的概念、精神和范围的情况下,可以改变所述方法以及本文所描述的方法的步骤或步骤序列。更具体地,将显而易见的是,化学和生理学相关的某些药剂可以取代本文所描述的药剂,同时将实现相同或类似的结果。对于本领域的技术人员来说显而易见的所有此类类似的替代和修改都被认为处于由所附权利要求限定的本发明的精神、范围和概念内。
- 基于主换热器系统的核反应堆功率主动调控方法及系统
- 一种基于灰度图像的核反应堆主泵故障辅助诊断系统及方法
