一种食管鳞癌血清蛋白生物标志物及其应用
文献发布时间:2024-07-23 01:35:21
技术领域
本发明属于生物标志物和分子生物学技术领域,具体提供一种食管鳞癌血清蛋白生物标志物及其应用。
背景技术
本发明背景技术中公开的信息仅仅旨在增加对本发明的总体背景的理解,而不必然被视为承认或以任何形式暗示该信息构成已经成为本领域一般技术人员所公知的现有技术。
食管癌是一种严重威胁人类健康的慢性非传染性疾病,依据其病理分型可以分为食管鳞状细胞癌(Esophageal Squamous-cell Carcinoma,ESCC)和食管腺癌(EsophagealAdenocarcinoma,EAC)。其中,食管鳞癌早期症状不明显,通常到中晚期才出现吞咽困难、声音嘶哑、饮水呛咳等明显症状。这导致很多食管鳞癌患者初诊时已为中晚期。早期和晚期食管鳞癌患者在治疗方式、不良反应、治疗费用、预后方面存在巨大差异,因此需要识别食管鳞癌高危人群并对其进行精准筛查,长期随访,以求在食管鳞癌的早期或食管高级别上皮内瘤变(以下简称癌前)阶段识别患者,改善食管鳞癌患者预后,降低食管鳞癌疾病负担。
生物标志物是一种可以被测量和评估的客观特征,是正常生理过程、疾病过程或对治疗干预的反应的指标,可以用作诊断工具,也可以用作疾病分期的工具。对食管鳞癌生物标志物的探索一般是通过对食管癌患者的组织或者体液,如血液、尿液等进行的。由于食管癌患者的组织样本一般是通过内镜检查得到的,而内镜检查是食管癌的诊断方式之一,所以组织不适合作为食管癌早筛生物标志物的探索材料。体液中的血液,在全身流动,在含有常见的血浆蛋白质的同时,也包含了如免疫球蛋白、非激素蛋白、组织蛋白等特殊物质。肿瘤细胞在其发生发展过程中会分泌或因细胞凋亡产生一些特殊的物质分布到外周血液中,机体也会因对肿瘤细胞产生反应而分泌一些特殊物质到外周血中。这些血液中的特殊物质能反应身体中癌症的发生发展情况。蛋白质是大多数细胞功能的直接执行者,也是当前大多数癌症治疗的药物靶点。对血液中蛋白生物标志物的探索,可能会在获得较好的诊断效能的同时,帮助识别肿瘤起源。所以需要通过研究血液中的蛋白质来探索食管鳞癌生物标志物。
然而,血浆蛋白质组的特点之一是各种蛋白浓度相差巨大,跨越12个数量级以上。处于高丰度端的蛋白质多为常见蛋白,如白蛋白,对疾病有诊断作用的蛋白质,多处于低丰度端。既往有学者基于血液样本使用蛋白质组学探索食管鳞癌血液蛋白标志物。然而,这些基于血液样本探索食管鳞癌生物标志物的研究存在一些不足。首先,这些研究的样本量普遍偏小,且鲜有外部验证。其次,大部分研究使用质谱技术对血液样本进行检测。受检测方法的影响,蛋白的重复性较差,且低丰度蛋白难以检测到。再次,大部分研究没有考虑食管鳞癌发病的地理差异,只纳入了高发区或者低发区的研究对象。最后,部分研究没有纳入早期患者,无法评价这些研究发现的生物标志物对早期患者的筛查效果。
此外,为了实现对一般人群进行食管鳞癌风险分层的目的,多位学者先后开发了多个食管癌筛查模型。这些模型按照变量的纳入情况可以大致分为两类,一类仅纳入危险因素,一类不仅纳入危险因素,还纳入了症状相关变量,如胸骨后疼痛,吞咽困难等。但这些仅基于危险因素构建的模型AUC值通常较低,不足0.80;添加症状变量后可以突破0.80。但食管癌一旦出现症状便已经到中晚期,此时再进行干预为时已晚。所以,需要尝试新的模型构建策略,在不加入描述症状的变量的前提下,提高预测模型区分病例和对照的能力,以获得更好的食管鳞癌风险分层效果。
发明内容
针对上述现有技术,本发明的目的在于提供一种食管鳞癌血清蛋白生物标志物及其应用。本发明以高发地区的研究对象为发现集,以低发地区的研究对象为验证集,使用能检测到血液中低丰度蛋白且重复性好的Olink蛋白组学技术探索并验证食管鳞癌血液蛋白标志物,并进一步使用logistic回归模型和机器学习算法基于危险因素和血液蛋白标志物构建及验证食管鳞癌预测模型。为我国食管鳞癌的诊断、早筛及一般人群风险分层工作供理论支持。基于上述研究成果,从而完成本发明。
具体的,本发明技术方案如下:
本发明的第一个方面,提供一种食管鳞癌血清蛋白生物标志物,所述食管鳞癌血清蛋白生物标志物为如下蛋白中的任意一个或多个:ABL1,ANXA1,CDKN1A,EGF,FADD,LYN,MetAP.2,SCAMP3,TGF.alpha,TXLNA,VIM,ADAM.TS.15,CXL17,ERBB2,GPC1,KLK13,RSPO3。
进一步的,所述食管鳞癌血清蛋白生物标志物为ADAM.TS.15,ANXA1,CDKN1A,GPC1,KLK13,RSPO3及TGF.alpha所组成的组。
本发明的第二个方面,提供检测上述生物标志物的试剂在制备筛查以及诊断食管鳞癌产品中的应用。
具体的,所述检测上述生物标志物的试剂为使用Olink蛋白组学技术检测时所采用的试剂。
所述筛查以及诊断食管鳞癌包括为对食管鳞癌的(辅助)早期筛查及(辅助)早期诊断。早期食管癌通常指病灶局限于黏膜内层的食管浸润性癌,无论有无区域淋巴结转移,一般对应食管高级别上皮内瘤变和I期食管癌患者。因此本发明中,早期食管鳞癌定义为癌前和I期食管鳞。
本发明的第三个方面,提供一种食管鳞癌风险预测系统,所述系统包括:
i)分析单元,其包含:用于确定受试者的待测样品中选自上述生物标志物表达水平的检测试剂,以及;
ii)评估单元,其包含:根据i)中确定的所述生物标志物表达水平判断所述受试者的患病情况。
其中,
所述分析单元中,待测样品为血液,进一步为血清。
所述检测试剂可以为使用Olink蛋白组学技术检测受试者血清蛋白时所采用的试剂。
所述评估单元中至少包含一个食管鳞癌预测模型,所述食管鳞癌预测模型为如下(a)或(b)中任意一种:
(a)基于上述生物标志物通过算法构建获得;
(b)基于危险因素通过算法构建的模型以及基于上述生物标志物通过算法构建的模型使用Stacking方法进行集成学习获得。
其中,所述(a)和(b)中,算法包括logistic回归模型、神经网络模型、随机森林模型和梯度提升决策树;
所述(b)中,所述危险因素包括年龄、性别、受教育程度、身高、牙齿缺失、吸烟、饮酒、饮热茶史以及癌症家族史中的任意一种或多种。
进一步的,所述食管鳞癌预测模型为基于危险因素中的logistic回归模型及随机森林模型和基于生物标志物中的神经网络模型及随机森林模型使用Stacking方法进行集成学习获得的。
所述食管鳞癌风险预测系统可用于对受试者食管鳞癌的(辅助)早期筛查及(辅助)早期诊断。
上述一个或多个技术方案的有益技术效果:
上述技术方案使用Olink蛋白组学技术检测了研究对象的血液,探索并验证了食管鳞癌的血液生物标志物,并对差异蛋白进行了GO富集分析、KEGG富集分析和蛋白质网络互作分析。并进一步基于危险因素和血液蛋白生物标志物,使用传统logistic回归模型和机器学习算法构建食管鳞癌预测模型。
上述技术方案为食管鳞癌的早期诊断筛查提供具有广泛适用性的血液蛋白生物标志物,同时上述技术方案中食管鳞癌预测模型的构建与验证将为一般人群中食管鳞癌风险分层工作提供支撑,因此具有良好的实际应用之价值。
附图说明
构成本发明的一部分的说明书附图用来提供对本发明的进一步理解,本发明的示意性实施例及其说明用于解释本发明,并不构成对本发明的不当限定。
图1为本发明实施例中发现集和验证集早期食管鳞癌患者与对照差异蛋白火山图;(A)发现集早期食管鳞癌患者和对照差异蛋白火山图;(B)验证集早期食管鳞癌患者和对照差异蛋白火山图。蓝色表示在病例组中表达降低,红色表示在病例组中表达上升,圆圈表示在发现集和验证集中均有差异且变化方向相同。
图2为本发明实施例中发现集和验证集中晚期食管鳞癌患者与对照差异蛋白火山图。其中,(A)发现集中晚期食管鳞癌患者和对照差异蛋白火山图;(B)验证集中晚期食管鳞癌患者和对照差异蛋白火山图。蓝色表示在病例组中表达降低,红色表示在病例组中表达上升,圆圈表示在发现集和验证集中均有差异且变化方向相同。
图3为本发明实施例中早期及中晚期食管鳞癌患者与对照差异蛋白韦恩图。
图4为本发明实施例中发现集及验证集食管鳞癌差异蛋白热图及单个蛋白AUC
(A)发现集食管鳞癌差异蛋白热图及单个蛋白AUC;(B)验证集食管鳞癌差异蛋白热图及单个蛋白AUC。
图5为本发明实施例中训练集和测试集中危险因素变量间的相关矩阵;(A)训练集危险因素变量间的相关矩阵;(B)测试集中危险因素变量间的相关矩阵。
图6本发明实施例中基于危险因素的食管鳞癌预测模型在测试集上的ROC曲线。
图7为本发明实施例中基于危险因素的食管鳞癌预测模型在测试集上的校准曲线。
图8为本发明实施例中训练集和测试集中血液蛋白生物标志物变量间的相关矩阵;其中,(A)训练集;(B)测试集。
图9为本发明实施例中LASSO回归结果;其中,A为LASSO回归交叉验证曲线;B为LASSO系数路径图。
图10为本发明实施例中基于生物标志物的食管鳞癌预测模型在测试集上的ROC曲线。
图11为本发明实施例中基于生物标志物的食管鳞癌预测模型在测试集上的校准曲线。
图12为本发明实施例中基于危险因素和生物标志物的食管鳞癌预测模型在测试集上的ROC曲线。
具体实施方式
应该指出,以下详细说明都是例示性的,旨在对本发明提供进一步的说明。除非另有指明,本文使用的所有技术和科学术语具有与本发明所属技术领域的普通技术人员通常理解的相同含义。
需要注意的是,这里所使用的术语仅是为了描述具体实施方式,而非意图限制根据本发明的示例性实施方式。如在这里所使用的,除非上下文另外明确指出,否则单数形式也意图包括复数形式,此外,还应当理解的是,当在本说明书中使用术语“包含”和/或“包括”时,其指明存在特征、步骤、操作、器件、组件和/或它们的组合。
现结合具体实例对本发明作进一步的说明,以下实例仅是为了解释本发明,并不对其内容进行限定。如果实施例中未注明的实验具体条件,通常按照常规条件,或按照试剂公司所推荐的条件;下述实施例中所用的试剂、耗材等,如无特殊说明,均可从商业途径得到。
实施例
材料和方法
1.研究设计
本研究中,使用的两个数据集均为病例对照研究设计。在生物标志物的发现与验证部分,使用研究对象来自食管鳞癌高发地区江苏泰兴的病例对照研究为发现集,以研究对象来自食管鳞癌低发地区山东济南周边的病例对照研究为验证集。在食管鳞癌预测模型构建与验证中,以发现集为训练集,以验证集为测试集。研究整体方案通过了山东大学齐鲁医院伦理委员会审查。所有研究均严格按照审批的方案进行,所有研究对象均签署知情同意书。
1.1发现集
2009年10月-2013年9月,本课题组在泰兴市中医院、泰兴市人民医院、泰兴市第二人民医院和泰兴市第三人民医院的胃镜室中建立了一个病例招募系统以便快速的招募病例。最终,在发现集中纳入了30名癌前患者、60名I期患者、70名II期患者和70名III/IV期患者。在泰兴市开展的病例对照研究在收集食管癌患者的同时,也收集胃癌患者。因食管癌和胃癌病例在年龄和性别分布上具有相似性,故将两种病例合并后按照性别和五岁年龄组在泰兴市人口登记系统中按频数匹配的方式随机抽取对照。因前期预调查应答率约为75%,故按1:1.3抽取对照。最终,本研究共纳入合格对照1992人,应答率为70%。在本研究中,进一步限制对照问卷信息完整、血液样本合格。按照性别和五岁年龄组与II期食管鳞癌患者进行频数匹配的方式,在符合条件的对照中随机抽取70例健康对照。
1.2验证集
验证集中的病例依托山东大学齐鲁医院消化系统肿瘤专病队列收集(自2022年10月份起开展),对照来源于山东省丘陵农村自然人群队列。在本研究中,进一步限制研究对象年龄为45-85岁、经病理诊断确定为食管鳞癌、既往无恶性肿瘤病史、血液样本合格且充足。符合额外纳入条件的研究对象共187例,其中,癌前患者49例,I期患者38例,II期患者37例,III/IV期患者63例。最终,通过随机抽样的方式从符合条件的患者中抽取癌前患者35例,I期患者35例,II期患者35例,III/IV期患者35例。考虑到山东大学齐鲁医院是国家卫生健康委委属(管)的三级甲等综合医院,来该院就诊的患者社会经济地位普遍较高且大多为济南市周边地区患者。为了尽可能的从病例的来源人群中抽取对照,本研究选择从山东省丘陵农村自然人群队列人群中的居住于城镇的人口中抽取对照。在符合研究纳入条件的研究对象中,按照2岁年龄组,性别相同与山东大学齐鲁医院消化系统专病队列中抽取的癌前及I期研究对象进行频数匹配,共纳入健康对照70例。
1.3检验效能
在本研究中,将92种蛋白质的假设检验的显著性水平设置为0.001,统计功效设置为0.90,患者和健康对照组显著生物标志物差异为标准差的0.8倍,通过PASS15软件计算得到每组需要样本量为69例。早期食管癌通常指病灶局限于黏膜内层的食管浸润性癌,无论有无区域淋巴结转移,一般对应食管高级别上皮内瘤变和I期食管癌患者。食管高级别上皮内瘤变为食管鳞癌的癌前病变。故在本研究中,将早期食管鳞癌患者定义为癌前和I期食管鳞癌患者,中晚期食管鳞癌患者定义为II期~IV期患者。在发现集中,健康对照70例,早期食管鳞癌患者90例,中晚期食管鳞癌患者140例。验证集中,健康对照70例,早期食管鳞癌患者70例,中晚期食管鳞癌患者70例。样本量满足统计学方法的要求。
1.4资料收集
1.4.1血液样本收集与储存
发现集和验证集研究对象血液样本的采集都严格按照标准进行。在采集血液样本前,要求受试者在至少空腹8小时,由经过专业培训的医护人员按照标准方法采集血液样本。
1.4.2问卷信息及查体信息收集
在发现集中,研究对象的基线信息是通过调查员使用电子问卷对受访者进行一对一访谈得到的。
在验证集中,病例的基线信息是通过查阅病历和在出院后对其进行电话随访得到的。对于符合纳入条件的、首次纳入的研究对象,通过查阅病历资料得到其人口学信息,如年龄、性别、身高等。待患者出院后,对于通过查阅出院记录和病理报告后确定纳入的研究对象,由经过统一培训的调查员使用纸质版问卷对其进行电话随访,收集其教育、吸烟、饮酒、饮茶、牙齿缺失、肿瘤家族史等信息。验证集中对照的基线信息中的年龄、性别、教育、牙齿缺失、吸烟、饮酒、饮热茶史、癌症家族史由调查员使用电子问卷对其进行一对一访谈得到。身高是由调查员对其进行体格检查得到的。
根据既往研究,整理研究对象的基线信息。一般人口学信息包括年龄(<55/55-65/65-75/>75)、性别(男性/女性)、受教育程度(小学及以下/初中及以上)、身高(男性:≤162/162-170/170-174/>174;女性:≤152/152-156/156-160/>160)。吸烟史定义为研究对象曾经在半年内每1-3天至少吸烟1支,否则为无吸烟史。有饮酒史定义为研究对象曾经在半年内每周至少饮酒1次,否则为无饮酒史。热茶定义为冲开后放置不足2分钟的茶水。饮热茶史定义为研究对象曾在6个月内每天至少饮用热茶一杯。研究对象的一级亲属中任意一个当前或曾经患有恶性肿瘤即定义为有癌症家族史。一级亲属指研究对象的父母,兄弟姐妹及子女。研究对象在成年之后有至少1颗牙齿被补过或缺失则定义为有牙齿缺失。
1.5实验室检测
本研究中,发现集和验证集的血液样本均是通过Olink蛋白组学技术进行检测的。与传统蛋白组学相比,Olink蛋白组学具有通量高、使用样品少、高灵敏性等多种优点。采用预处理标准化程序进行数据预处理。仪器下机后导出文件后缀为“XXX.q100”的文件。打开Olink NPX Signature 1.5.3.0软件,导入该文件,设置试剂panel信息,试剂版本号、样本名称,点击“OK”,软件自动分析后输出NPX.Excel文档和QC AR.PDF文档。NPX.Excel为样本蛋白检测结果,QC AR.PDF文档为质控报告。输出的蛋白检测结果为标准化蛋白质表达值(NPX值),是由qPCR的CT值经过对数转换后得到的相对定量单位,相差1代表相差两倍。该值可以直接用来进行统计分析。本研究使用肿瘤II panel进行样本检测,该panel检测的92种蛋白质名称如下表1。
表1检测的蛋白标志物名称
1.6统计学分析
本研究所有的统计分析与结果展示,除特殊说明外,均采用R(v4.2.2)完成等。所有的统计学假设检验均为双侧检验,检验水准(α)设为0.05。
1.6.1基线特征描述性统计分析
对发现集和验证集中的食管鳞癌患者和对照的年龄、性别、生活习惯等基线特征进行统计学描述,并进行组间差异检验。对于计数资料,使用频数和百分比(%)进行统计描述,并依据数据情况选择卡方检验或Fisher精确概率法来进行组间比较分析。对于计量资料,首先使用Shapiro-Wilk检验对其进行正态性检验。对于符合正态分布的计量资料使用均数±标准差(Mean±SD)进行描述,使用t检验进行组间比较;对于不符合正态分布的计量资料,使用中位数(下四分位数,上四分位数)进行描述,组间比较实用Wilcoxon秩和检验。
1.6.2差异表达蛋白确定
首先使用Shapiro-Wilk法对各个蛋白的表达量进行正态性检验。对符合正态分布的蛋白,使用t检验进行组间比较;对不符合正态分布的蛋白表达量,使用Wilcoxon秩和检验进行组间比较,采用Benjamini-Hochberg方法调整P值,以控制错误发现率。使用“ggplot2”等包绘制火山图。将在发现集和验证集的病例和对照间均存在显著差异且变化方向相同的蛋白作为差异蛋白。
对筛选出的差异蛋白做基因本底(Gene Ontology,GO)功能分析和京都基因和基因组百科(Kyoto Encyclopedia of Genes and Genomes,KEGG)通路富集分析,使用“enrichplot”包进行可视化。使用相互作用基因/蛋白检索工具(Search tool for theretrieval of interacting genes/proteins,STRING)(http://string-db.org)探索差异蛋白间的蛋白质相互作用网络(Protein-protein interaction networks,PPI)。互作网络分析以STRING人类数据库为背景数据集。
1.6.3差异蛋白热图及区分能力初探
使用“pheatmap”包基于确定的差异蛋白在发现集和验证集中绘制差异蛋白热图,并分别对差异蛋白进行聚类。为了初步探索差异蛋白区分病例和对照的能力,使用非条件logistic回归在发现集和验证集中,对每一个蛋白进行回归,并通过计算AUC比较差异蛋白区分病例和对照的能力。
1.6.4预测模型的构建、验证与评价
本部分使用的变量有年龄、性别、受教育程度、身高、牙齿缺失、吸烟、饮酒、饮热茶史、癌症家族史和确定的食管鳞癌生物标志物,即ABL1、ANXA1、CDKN1A、EGF、FADD、LYN、MetAP.2、SCAMP3、TGF.alpha、TXLNA、VIM、ADAM.TS.15、CXL17、ERBB2、GPC1、KLK13、RSPO3。
1.6.4.1数据预处理
为了消除量纲的影响,对连续变量进行了标准化处理。对分类变量进行了哑变量处理。为了避免变量间多重共线性和冗余对模型构建的影响,采用R中的“glmnet”包采用最小绝对收缩和选择算法(Least absolute shrinkage and selection operator,LASSO)对变量进行筛选。LASSO回归,也被称作套索回归,是一种线性回归的缩减方法,通过构造一个惩罚函数来将回归模型中的变量系数进行压缩,将某些不重要的系数压缩到0,从而进行变量选择,来达到防止过度拟合,解决共线性问题的目的。在本研究中,以Lambda.1se为阈值,将病例与对照作为二分类结局变量,识别关键变量,并将关键变量纳入下一步分析中。
1.6.4.2预测模型构建
模型在训练集上的构建过程如下:超参数的调整采用三次五折交叉验证,通过网格搜索策略进行。将训练集数据随机五等分为A1-A5,依次从A1-A5中选择一个作为验证集,余下子集作为训练集。在训练集上使用网格搜索策略训练模型,在验证集上验证模型的预测效果。当每一个子集都被选做一次验证集后,视为完成一次五折交叉验证。三次五折交叉验证即将上述过程重复三次。本部分模型的评价标准为验证集上平均AUC最高,并将该参数作为最优参数。使用最优参数在整个训练集上重新训练模型作为最终模型。
本部分使用R软件中的“caret”包进行模型的构建与预测。该包为R中200多个包提供了统一的接口,可以通过trainControl函数、train函数实现设置模型训练方法、模型挑选方法等功能。
本研究先基于危险因素和生物标志物分别采用logistic回归模型、神经网络模型、随机森林模型和梯度提升决策树模型构建预测模型,然后使用基于危险因素构建的最优的2个模型、基于生物标志物两种策略下最优的2个模型使用Stacking方法进行集成学习以获得预测效果更好的模型。
本部分研究采用R软件中的“nnet”包(version 7.3-19)进行神经网络模型的分析,训练过程中调节的参数是隐藏层节点数(Hidden units,size)和权值衰减(Weightdecay,decay)。隐藏层节点数与网络的复杂程度相关。
本部分研究采用R软件中的“randomForest”包(version 4.7-1.1)进行随机森林模型的构建,训练过程中调节的参数是决策树数目(ntree)和输入特征(mtry)。决策树数目指森林中决策树的数量,一般情况下,树的数量越多越好,但随着树的数量的增加,计算时间也会增加。输入特征指每个节点上随机选择的特征数量,较小的输入特征值会增加树之间的差异性,但可能会降低模型的准确性,较大的输入特征值则会增加模型的准确性,但有可能导致过拟合。
本部分研究采用R软件中的“gbm”包(version 2.1.8.1)进行梯度提升决策树模型的构建。学习率(shrinkage),决策树数目(n.trees),树的最大深度(interaction.depth)和叶节点最小观测数(n.minobsinnode)是在训练过程中需要调整的参数。学习率是指每一个决策树在每次迭代中对最终模型的贡献程度,介于0-1之间,用于控制每一步的缩减量。如果学习率较小,则每个决策树的贡献较小,需要的决策树数目则越多,模型的训练速度会变慢,但可能会得到泛化性能更好的模型。相反,学习率较高,则会加快模型训练速度,可能导致过拟合。一般情况下,学习率和决策树数目相互依赖,是梯度提升决策树模型两个最重要的参数。树的最大深度与叶结点最小观测数控制决策树的复杂度。Stacking集成模型是机器学习集成模型的一种,通常将一个或多个基学习器与元学习器堆叠在一起以获得更好的预测效果。其基本构建方法是,首先基于原始数据构建基于不同算法的多个基学习器,然后将基学习器的输出值作为新的特征值训练元学习器。通常情况下,第一层模型中会选择拟合程度较高的模型,第二层则倾向使用较简单的模型,如logistic回归等。本研究使用了神经网络、随机森林等机器学习模型基于危险因素及生物标志构建模型,分别选择基于危险因素和生物标志物模型中AUC值最大的前两个模型构建Stacking集成模型,为避免过度拟合,使用了logistic回归模型。
1.6.4.3类不平衡问题
在本研究中,病例数大于对照数,存在不平衡分类问题。本研究尝试基于“ROSE”包采用过采样技术、欠采样技术和合成少数采样技术对数据集进行处理。但处理后模型在测试集的效能未见明显改善,因此仍选择按照原结果进行汇报。
1.6.4.4预测模型评价
本研究中构建的食管鳞癌预测模型,最终输出的为发生食管鳞癌的概率,取值为0~1。选取训练集上最大F1值处作为概率值的预警值,小于该值判定为未患食管鳞癌,大于该值判定为患食管鳞癌。基于此预警值,进一步评价预测模型的预警性能。所用的评价指标如下:
混淆矩阵(Confusion matrix),同时被称为可能性矩阵或可能性表格。以预测分类结果为横标目,以实际分类结果为纵标目,以四格表的形式直观反映模型分类的结果。在本研究中,其基本模式如见表2。
准确率(Accuracy):指被正确分类的食管鳞癌患者占所有研究对象的比例。计算公式为(TP+TN)/(TP+FP+TN+FN)。取值为0~1,准确率取值越大,分类效果越好。
灵敏度(Sensitivity):又称为召回率、真阳性率,指被正确预测为食管鳞癌的患者占全部食管鳞癌患者的比例,计算公式为(TP)/(TP+FN)。
特异度(Specificity):又称为真阴性率,指未患食管癌的研究对象被正确预测的比例,计算公式为TN/(TN+FP)。
表2混淆矩阵基本模式
阳性预测值(Positive predictive value):又称为精确率,指预测为食管鳞癌的患者中真正食管鳞
癌患者所占的比例,计算公式为TP/(FP+TP)。
阴性预测值(Negative predictive value):指预测为非食管鳞癌的研究对象中真正为非食管鳞癌患者所占的比例,计算公式为TN/(FN+TN)。
F1值:灵敏度与阳性预测值的调和平均数,计算公式为2TP/(2TP+FP+FN)。
受试者工作特征曲线((receiver operating characteristic curve,ROC)及AUC值:ROC曲线以假阳性率为横坐标,以真阳性率为纵坐标。通过分类阈值由小至大,将样本依据不同阈值分类并计算假阳性率与真阳性率绘制而成。AUC值为ROC曲线下面积,是目前公认的模型预测性能的良好评价指标之一。AUC值越大,模型预测性能越好,通常认为AUC值≥0.80时模型的预测性能良好,AUC值≥0.90时模型的预测性能优异。
1.6.4.5变量相对重要性
对于每一个单模型,使用“caret”包输出每一个变量的相对重要性并对其进行排序,以帮助识别重要的变量。对于Stacking集成模型,由于该模型是集成其他模型得到的,第二层学习器不再使用原始数据,故Stacking集成模型未进行变量重要性排序。
1.7敏感性分析
为了排除标warning样本对生物标志物发现及验证的潜在影响,本研究通过将发现集及对照集中标waring的样本进行排除分析来进行敏感性分析。
1.8质量控制
本研究在课题设计、调查实施、实验室检测及数据分析阶段均采取了均采取相应的措施进行质量控制,以减少潜在的偏倚。
2.研究结果
2.1研究对象基本信息
本研究发现集中共纳入70例对照和230例病例。验证集中有1例对照因血清样本中脂质过多被排除,故纳入69例对照和140例病例。研究对象的基本人口学特征和癌症分期结果见表3。
表3发现集及验证集研究对象基本信息
*各组之和不等于总数是因为存在缺失值。
2.2差异表达蛋白质筛选
在发现集中,早期食管鳞癌患者中有40个蛋白质与对照组有明显差异,其中上调的蛋白有14个,下降的蛋白有26个。在验证集中,早期食管鳞癌患者有36个蛋白与对照组差异明显,其中上调的蛋白18个,下降的蛋白18个。在发现集和验证集中均差异表达且变化方向相同的蛋白一共有21个,分别是ABL1,ANXA1,CDKN1A,EGF,FADD,LYN,MetAP.2,SCAMP3,TGF.alpha,TXLNA,VIM,ADAM.TS.15,CXL17,ERBB2,FCRLB,GPC1,KLK13,LYPD3,RSPO3,TNFRSF6B,TRAIL。详见图1。
在发现集中,中晚期食管鳞癌患者中有53个蛋白质与对照组有明显差异,其中上调的蛋白有18个,下降的蛋白有35个。在验证集中,中晚期食管鳞癌患者有50个蛋白与对照组差异明显,其中上调的蛋白34个,下降的蛋白16个。在发现集和验证集中均差异表达且变化方向相同的蛋白一共有27个,分别是ABL1,ANXA1,CDKN1A,EGF,FADD,GZMH,IL6,LYN,MetAP.2,S100A11,SCAMP3,TGF.alpha,TXLNA,VEGFA,VIM,ADAM.TS.15,CD160,CPE,CXL17,ERBB2,ERBB4,ESM.1,GPC1,ITGAV,KLK13,PODXL,RSPO3。详见图2。
对差异蛋白功能进行KEGG富集分析和GO富集分析,表明相比于对照,早期食管鳞癌患者受影响较大的KEGG通路有ErbB信号通路(ErbB signaling pathway)、非小细胞肺癌癌症(Non-small cell lung cancer)、癌症胰腺癌(Pancreatic cancer)、前列腺癌症(Prostate cancer)、EB病毒感染(Epstein-Barr virus infection)、癌症膀胱(Bladdercancer)、子宫内膜癌症(Endometrial cancer)、铂类药物耐药性(Platinum drugresistance)、胶质瘤(Glioma)、EGFR酪氨酸激酶抑制剂耐药性(EGFR tyrosine kinaseinhibitor resistance)等;中晚期食管鳞癌患者受影响较大的通路有Pl3K-Akt信号通路(Pl3K-Akt signaling pathway)、ErbB信号通路(ErbB signaling pathway)、癌症中的蛋白聚糖(Proteoglycans in cancer)、癌症胰腺癌(Pancreatic cancer)、EGFR酪氨酸激酶抑制剂耐药性(EGFR tyrosine kinase inhibitor resistance)、HlF-1信号通路(HlF-1signaling pathway)、卡波西肉瘤相关疱疹病毒感染(Kaposi sarcoma-associatedherpesvirus infection)、癌症膀胱(Bladder cancer)、非小细胞肺癌癌症(Non-smallcell lung cancer)、前列腺癌症(Prostate cancer)。GO富集分析表明,在生物过程层面,早期食管鳞癌差异蛋白主要富集在淋巴细胞增殖(lymphocyte proliferation)、胶质发生(gliogenesis)、单核细胞增殖(mononuclear cell proliferation)、ERBB2信号通路(ERBB2 signaling pathway)、白细胞增殖(leukocyte proliferation)等通路;中晚期食管鳞癌差异蛋白主要富集在细胞粘附的正调控(positive regulation of celladhesion)、肽基酪氨酸磷酸化的阳性反应(positive regulation of peptidyl-tyrosinephosphorylation)、ERBB2信号通路(ERBB2 signaling pathway)、细胞间粘附的调控(regulation of cell-cell adhesion)等通路。
在细胞组分层面,早期食管鳞癌差异蛋白主要在内吞囊泡膜、细胞基膜(basalplasma membrane)、细胞基底部(basal part of cell)等位置;中晚期食管鳞癌差异蛋白主要在囊泡、皱褶(ruffle)、粘附体接合(adherens junction)等处。
在分子功能层面,早期食管鳞癌差异蛋白主要涉及胞外基质结合(extracellularmatrix binding)、跨膜受体蛋白酪氨酸激酶激活剂作用(transmembrane receptorprotein tyrosine kinase activator act)、蛋白激酶激活剂活性(protein kinaseactivator activity)、激酶激活因子(kinase activator activity)、层粘连蛋白结合(laminin binding)等;中晚期食管鳞癌差异蛋白主要涉及生长因子受体结合(growthfactor receptor binding)、胞外基质结合(extracellular matrix binding)、蛋白酪氨酸激酶激活剂活性(protein tyrosine kinase activator activity)、表皮生长因子受体结合(epidermal growth factor receptor binding)、蛋白激酶激活剂活性(proteinkinase activator activity)等。
对早期食管鳞癌差异蛋白和中晚期差异蛋白进行PPI分析,结果显示无论早期还是中晚期食管鳞癌差异蛋白中均存在显著的蛋白质互作网络(早期食管鳞癌:PPlenrichment P-value:3.87e-06;中晚期食管鳞癌:PPl enrichment P-value:2.39e-09)。使用GO富集分析对具有相互作用的蛋白进行分析,在早期食管鳞癌中,这些蛋白质最显著富集的三个生物学过程胶质生成、ERBB2-EGFR信号通路、表皮生长因子受体信号通路;在晚期食管鳞癌中,最显著富集的三个生物学过程为细胞群增殖的正调控、表皮生长因子受体信号通路、细胞群增殖的调控。
2.3生物标志物的确定及其诊断价值
因本研究的目的是希望找到能区分病例和对照的血液蛋白生物标志物,所以,将对照与早期、对照与中晚期的蛋白取交集后作为经验证的生物标志物,分别是ABL1,ANXA1,CDKN1A,EGF,FADD,LYN,MetAP.2,SCAMP3,TGF.alpha,TXLNA,VIM,ADAM.TS.15,CXL17,ERBB2,GPC1,KLK13,RSPO3。详见图3。
在发现集和验证集使用生物标志物绘制热图,无论是在发现集中还是在验证集中,病例组和对照组均呈现明显的差异,且差异在验证集中更为显著。在蛋白聚类方面,差异蛋白在发现集和验证集中聚类大致相同。无论是在发现集还是验证集,同一个差异蛋白区分早期患者和中晚期患者的能力均大致相当。在发现集中,AUC的取值范围是0.61-0.80,区分病例和对照能力最强的蛋白质是ANXA1。在验证集中,ROC曲线下面积的取值范围是0.64-0.98,区分能力最强的蛋白质是ABL1。详见图4。
2.4基于危险因素构建食管鳞癌预测模型
2.4.1纳入模型的预测变量筛选
将本课题组既往发现的与食管鳞癌发病相关的变量纳入,用于构建基于危险因素的食管鳞癌预测模型。为避免多重共线性对模型构建的影响,通过计算变量间相关矩阵来检验变量间的相关性。图5展示预测变量间的相关性分析结果。总体来看,无论在训练集还是测试集,均未发现相关性较高的危险因素。所以使用全部危险因素变量构建模型。
2.4.2预测模型构建
在训练集上,通过三次五折交叉验证、参数调优后,神经网络模型、随机森林模型和梯度提升决策树模型的最优超参数如所表4所示。
2.4.3模型预测能力表现
本部分研究基于传统logistic回归和神经网络、随机森林、梯度提升决策树三种机器学习算法构建食管鳞癌预测模型。四种模型中在测试集上的预测效果均一般。四种模型在测试集上的AUC值最高为73.40%,准确度最高为70.33%。四个模型在训练集和测试集上的各预测效果评价指标见表5。在测试集上的ROC曲线及相应AUC值(95%CI)见图6。
表4基于危险因素的食管鳞癌预测模型最优超参数
表5基于危险因素的食管鳞癌预测模型在训练集和测试集上的预测效果评价
2.4.4校准曲线
模型的校准曲线以预测概率为横坐标,以实际概率为纵坐标。从图中可以看出,logistic回归模型的校准曲线与45度对角线较为贴合,梯度提升决策树模型和神经网络模型在实际风险较低时(<50%)会低估风险。见图7。
2.4.5预测变量相对重要性排序
表6展示了logistic回归模型、神经网络、随机森林模型和梯度提升决策树模型中各预测变量的相对重要性排序。以logistic回归模型为例,最重要的三个变量分别是身高、肿瘤家族史和饮酒。这三个变量在随机森林模型、梯度提升决策树模型预测变量相对重要性排序中均排在前四。从四个模型来看,身高是最重要的变量,logistic回归模型、随机森林模型及梯度提升决策树模型中均排到了最重要的位置。
表6基于危险因素的食管鳞癌预测模型预测变量相对重要性排序
2.5基于蛋白生物标志物构建食管鳞癌预测模型
2.5.1纳入模型的变量筛选
将发现的差异蛋白作为蛋白生物标志物纳入分析,构建基于生物标志物的食管鳞癌预测模型。为了避免变量冗余对模型的影响,使用相关矩阵检验变量间的相关性。从图中可以发现,一些蛋白间相关性较大,如ABL1与SCAMP3,在训练集和测试集中相关性系数均超过0.9,详见图8。所以需要使用LASSO回归进行变量选择。LASSO回归的结果见图9。以Lambda.1se作为阈值,留下的变量为ADAM.TS.15,ANXA1,CDKN1A,GPC1,KLK13,RSPO3及TGF.alpha。
2.5.2预测模型构建
在训练集上,通过三次五折交叉验证、参数调优后,神经网络模型、随机森林模型和梯度提升决策树模型的最优超参数如表7所示。
表7基于生物标志物的食管鳞癌预测模型最优超参数
2.5.3模型预测能力表现
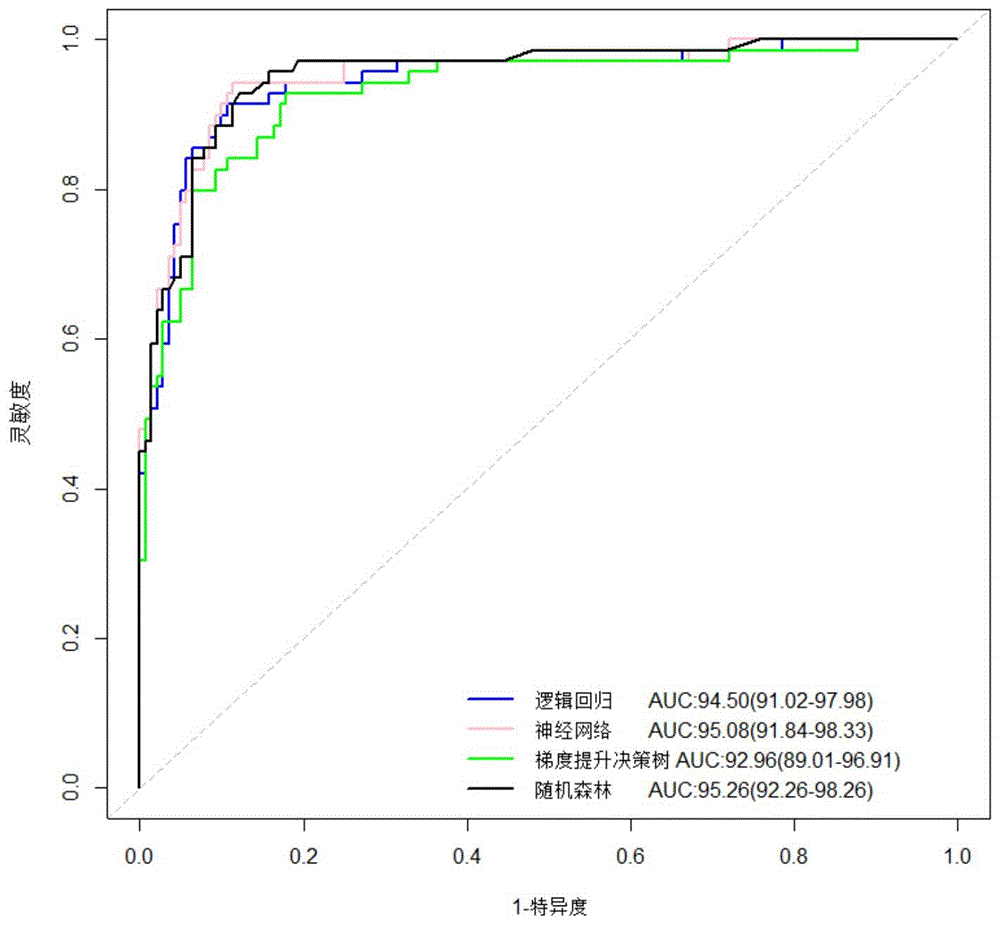
本部分研究基于生物标志物使用传统logistic回归模型和神经网络、随机森林、梯度提升决策树三种机器学习算法构建食管鳞癌预测模型。四种模型中在测试集上的预测效果均较好,测试集上的AUC值均超过92%,准确度均超过86%。四个模型在训练集和测试集上的各预测效果评价指标见表8。在测试集上的ROC曲线及相应AUC值(95%CI)见图10。
表8基于血液生物标志物的食管鳞癌模型在训练集和测试集上的预测效果评价
2.5.4校准曲线
从图11中可以看出,logistic回归模型、神经网络模型和梯度提升决策树模型的校准曲线在实际风险较低时(<40%)与45度对角线贴合较为紧密,在风险较高时(>40%)则倾向高估风险。随机森林模型则一直倾向于高估风险。
2.5.5预测变量相对重要性排序
表9展示了基于生物标志物的logistic回归模型、神经网络、随机森林模型和梯度提升决策树模型中各预测变量的相对重要性排序。Logistic回归模型和神经网络模型在变量重要性排序中相近,重要性排名前三的变量都是TGF.alpha、RSPO3及CDKN1A。随机森林和梯度提升决策树模型的变量重要性排名则比较一致,重要性排名前五的变量分别是ANXA1、RSPO3、TGF.alpha、CDKN1A、ADAM.TS.15。
表9基于危险因素的食管鳞癌预测模型预测变量相对重要性排序
2.6基于危险因素及蛋白生物标志物构建食管鳞癌预测模型
Stacking集成模型是基于危险因素中的logistic回归模型及随机森林模型和基于蛋白生物标志物中的神经网络模型及随机森林模型构建的。在测试集上Stacking模型的AUC为94.99%(92.02%,94.99%),其ROC曲线见图12。在训练集中Stacking模型的最大F1值为97.70%,对应的概率值为0.84。以此为界值,在测试集上评价模型的效果分类,此时混淆矩阵如表11所示。在测试集上,模型准确度为91.87%(87.36%,94.86%),灵敏度为93.57%(88.23%,96.58%),特异度为88.41%(78.75%,94.01%),阳性预测值为94.24%(89.05%,97.06%),阴性预测值为87.14%(77.34%,93.09%),F1值为93.91%。
表10基于危险因素和生物标志物的Stacking集成模型在训练集和测试集上的预测效果评价
表11基于危险因素和生物标志物的Stacking集成模型在测试集上对应的混淆矩阵
总之,本研究基于在江苏省泰兴市开展的一项基于人群的病例对照研究、山东大学齐鲁医院消化系统肿瘤专病队列和山东省丘陵农村自然人群队列,构建了两项病例对照研究,以研究对象来源于食管鳞癌高发地江苏泰兴的病例对照研究为发现集,以研究对象来自食管鳞癌低发地区山东济南周边的病例对照研究为验证集,使用Olink蛋白组学技术检测了研究对象的血液,探索并验证了食管鳞癌的血液生物标志物,并对差异蛋白进行了GO富集分析、KEGG富集分析和蛋白质网络互作分析。并进一步基于危险因素和血液蛋白生物标志物,使用传统logistic回归模型和机器学习算法构建食管鳞癌预测模型。
主要结论如下:
(1)经验证的食管鳞癌血液差异蛋白共有17个,分别是ABL1,ANXA1,CDKN1A,EGF,FADD,LYN,MetAP.2,SCAMP3,TGF.alpha,TXLNA,VIM,ADAM.TS.15,CXL17,ERBB2,GPC1,KLK13,RSPO3。这些蛋白质可以在一定程度上区分病例和对照,AUC曲线下面积最低为0.61,最高为0.98。本研究结果将为食管鳞癌的早期诊断筛查提供具有广泛适用性的血液蛋白生物标志物。
(2)基于危险因素使用传统logistic和机器学习构建的预测模型在测试集上的AUC一般为70%左右;基于血液生物标志物构建的4个预测模型在测试集上的AUC均大于92%;基于危险因素和生物标志物构建的Stacking集成模型在测试集上的AUC为96.26%。身高、肿瘤家族史、饮酒、ANXA1、TGF.alpha、CDKN1A、RSPO3是食管鳞癌的重要预测因子。食管鳞癌预测模型的构建与验证将为一般人群中食管鳞癌风险分层工作提供支撑。
上述实施例只为说明本发明的技术构思及特点,其目的在于让熟悉此项技术的人士能够了解本发明的内容并据以实施,并不能以此限制本发明的保护范围。凡根据本发明精神实质所作的等效变化或修饰,都应涵盖在本发明的保护范围之内。
- 一种用于食管鳞癌早期筛查和诊断的血清蛋白标志物、试剂盒及检测方法
- 一种食管鳞癌外泌体富含的miRNA作为诊断食管鳞癌的标志物中的应用
