基于DNA-铜纳米簇与HCR信号放大的CRISPR/Cas12a检测系统及其应用
文献发布时间:2024-04-18 19:58:53
技术领域
本发明涉及生物检测,具体涉及基于DNA-铜纳米簇与HCR信号放大的CRISPR/Cas12a检测系统及其应用。
背景技术
杂交链式反应(HCR)是一种新型无酶参与的核酸聚合反应,仅由目标分子引发若干种自身稳定的DNA茎环结构发生级联反应产生具有切口的长链DNA结构,该反应属于等温扩增技术,仪器操作简单,且该技术不需要辅助酶的参与,反应条件温和,反应的重现性好。除外,DNA-铜纳米簇是一种能以DNA为模板进行即时合成的荧光纳米材料,具有价格低廉、合成简单、生物相容性好等优点。CRISPR/Cas12a检测技术仅需一段RNA和CRISPR相关蛋白(Cas)组成二元复合物就可发挥识别目标和信号放大功能,具有操作简单、特异性强、反应迅速等特点。然而,现有的CRISPR/Cas12a检测技术的荧光基团修饰报告子具有价格昂贵、合成复杂、稳定性不足等缺点,导致检测系统灵敏度降低,检测误差较大。因此,很有必要开发出价格低廉、易于合成兼顾荧光性能的报告子。
发明内容
发明目的:本发明的目的在于提供一种操作简单、成本低廉、灵敏度高、特异性强的基于DNA-铜纳米簇与HCR信号放大的CRISPR/Cas12a检测方法。本发明的另一目的是提供一种基于DNA-铜纳米簇与HCR信号放大的CRISPR/Cas12a检测方法在猴痘病毒核酸检测中的应用。
技术方案:本发明所述的基于DNA-铜纳米簇与HCR信号放大的CRISPR/Cas12a检测系统,所述检测系统包括DNA-铜纳米簇体系、HCR反应体系、CRISPR/Cas12a识别体系;所述HCR反应体系包括启动链inDNA、两种发夹DNA以及沉默链siDNA,其中所述启动链inDNA包含触发HCR的序列;siDNA具备结合HCR反应体系中的启动链inDNA的能力,用于沉默HCR反应;当分析的目标核酸存在时,激活CRISPR/Cas12a识别体系,不断切割环境中的沉默链siDNA,结合启动链inDNA的siDNA被切割后,启动链inDNA触发HCR反应,使两种稳定共存的发夹DNA打开后,交替互补形成具有切口的长链DNA,该长链DNA上规律分布poly(AT-TA),铜原子聚集在富含poly(AT-TA)的DNA链上形成DNA-铜纳米簇,发出粉红色荧光,进行荧光检测。
所述的基于DNA-铜纳米簇与HCR信号放大的CRISPR/Cas12a检测系统,所述CRISPR/Cas12a识别体系由Cas12a与crRNA在反应缓冲液中混合后孵育而得。
基于DNA-铜纳米簇与HCR信号放大的CRISPR/Cas12a检测方法,包括以下步骤:
(1)配制crRNA-Cas12a二元复合物:将Cas12a、crRNA在反应缓冲液中混合,放置冰水浴上备用;
(2)制备待测目标核酸Target:分别将Target-1和Target-2干粉离心后使用水溶解,混合均匀,制成溶液。分别取同体积的Target-1和Target-2混合均匀,置于PCR仪中升温至85-105℃保持2-15min,之后每隔1-5min温度下降3-8℃,直到降至10-15℃,即可制得双链杂交的目标核酸溶液,存储于3-8℃备用。
(3)将不同浓度的目标核酸溶液、待测样品溶液分别与crRNA-Cas12a二元复合物混合,再加入siDNA,混合溶液于30-40℃孵育,孵育结束后,升温,70-120℃灭活Cas12a,然后加入inDNA进行链杂交,再分别添加H
(4)吸取步骤(3)的溶液进行荧光检测;
(5)绘制目标核酸的标准曲线图,并计算待测样品的浓度。
所述的检测方法,DNA-铜纳米簇由硫酸铜在抗坏血酸钠还原下由二价铜离子转变为铜原子聚集在富含poly(AT-TA)的双链DNA片段上形成。
所述的检测方法,所述Target-1和Target-2为互补链。
所述的检测方法,所述inDNA的序列为:CAGCGGGGTATATAACGGCAGGCCCACGGC,H
当目标核酸为猴痘病毒核酸时,Target-1、Target-2的序列选自:TTTGATGATGTTATTCCGGTTAA、TTAACCGGAATAACATCATCAAA。
所述的检测方法,步骤(1)中所述反应缓冲液包括NaCl、Tris-HCl、MgCl
所述的检测方法,所述反应缓冲溶液中NaCl、Tris-HCl、和MgCl
所述的检测系统或所述的检测方法在DNA病毒核酸检测中的应用。
所述的检测系统或所述的检测方法在猴痘病毒、牛痘病毒、天花病毒、非洲猪瘟病毒或弓形虫的核酸检测中的应用。
本发明中Cas12a核酸酶活性的激活依赖一段与向导RNA互补的DNA,该DNA即目标检测核酸。这段DNA在启动Cas12a之后,使得Cas12a开始对环境中存在的siDNA不断切割,该siDNA即启动链DNA的沉默DNA。环境中的siDNA被切割,使得启动链DNA可以顺利触发HCR,产生DNA-铜纳米簇,发出粉红色荧光,从而能够实现相应目标物的检测。
本发明的原理见附图1,Cas12a与crRNA在溶液中组装成crRNA-Cas12a二元复合物,加入与启动链inDNA互补的沉默siDNA,当分析的目标核酸存在时,可以激活Cas12a,切割环境中的沉默siDNA,这时添加启动链inDNA、H1和H2可触发HCR反应,产物富含poly(AT-TA)的重复序列与还原Cu
本发明将DNA-铜纳米簇与HCR信号放大与CRISPR/Cas12a结合,摒弃了原有CRISPR/Cas12a报告技术价格昂贵、合成复杂、稳定性不足的缺点,DNA-铜纳米簇形成的条件温和,反应迅速,合成简单,荧光性能好。而HCR能显著地放大信号,反应条件温和,由熵驱动催化,不需要其他酶参与,在该体系中发挥了信号二次放大的作用。
有益效果:与现有技术相比,本发明具有以下显著优点:
(1)本发明基于DNA-铜纳米簇与HCR信号放大的CRISPR/Cas12a报告技术灵敏度高、特异性好、检测限低,可以准确地检测病毒核酸。(2)本发明的crRNA具有可编辑性,能够根据检测对象的特征核酸进行设计,所设计的crRNA与致病核酸在一定序列要求下进行碱基互补,就能诱导Cas12a激活,从而实现其他致病核酸的检测。(3)HCR能显著地放大信号,反应条件温和,由熵驱动催化,不需要其他酶参与,还消除了复杂的热循环步骤,实现了Cas12a对抑制链的切割效果。(4)本发明基于DNA-铜纳米簇与HCR信号放大的CRISPR/Cas12a报告技术可用于猴痘病毒核酸质粒的检测,相较于传统的检测方法,该报告技术所涉及到的CRISPR/Cas12a、HCR反应体系、DNA-铜纳米簇、可在常温条件下进行,反应条件温和,反应迅速,检测灵敏度高、特异性强、设备要求低,为猴痘病毒的临床检测提供了一个有潜力的可靠工具。
附图说明
图1是基于DNA-铜纳米簇与HCR信号放大的CRISPR/Cas12a报告技术的原理图;
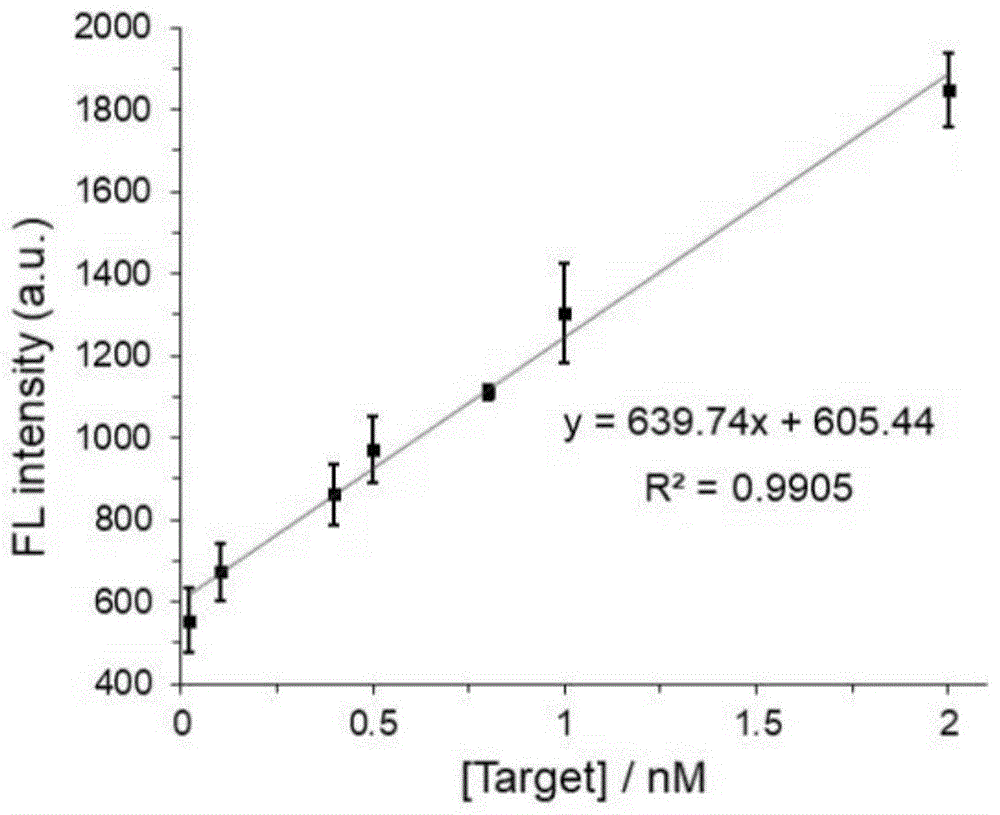
图2是不同浓度Target序列检测荧光所得到的线性;
图3是不同种类病毒特征核酸片段检测荧光结果;
图4是不同浓度猴痘病毒特征核酸质粒检测荧光结果;
图5是不同种类病毒特征核酸质粒检测荧光结果。
具体实施方式
以下结合附图和实施例对本发明做进一步说明。
实施例中所述实验方法,如无特殊说明,均为常规方法。药品和试剂:实验中使用的DNA、crRNA均由生工生物工程(上海,中国)合成,并且经HPLC纯化。LbCas12a购买自无锡市江原实业技贸有限公司。抗坏血酸钠、CuSO
实施例1
基于DNA-铜纳米簇与HCR信号放大的CRISPR/Cas12a报告技术检测Target
(1)配制crRNA-Cas12a二元复合物:将50nMCas12a、50nMcrRNA在反应缓冲液中(50nMNaCl、10mMTris-Hcl、10mM MgCl2、0.1g/mL牛血清蛋白,pH 7.9)混合,放置冰水浴上备用。
(2)制备待测目标核酸Target:分别将Target-1和Target-2干粉12000rpm离心2min后使用ddH
(3)稀释上一步骤所得的Target溶液至多种不同浓度,将2μLTarget(0.5nM、2.5nM、5nM、7.5nM、10nM、12.5nM、20nM、25nM和50nM)与50nMcrRNA-Cas12a二元复合物混合,再加入0.5μMsiDNA,混合溶液于37℃孵育1h。孵育结束后,升温至90℃灭活Cas12a。然后加入0.5μMinDNA进行链杂交,再分别添加1μMH
(4)吸取步骤(3)的溶液进行荧光检测,采用BioTek Synergy-H1多功能酶标仪检测,选用384孔检测荧光终点学检测模式,检测条件为:Ex:340nm,Em:600nm。如图2所示,为该报告技术用于猴痘病毒Target序列的标准曲线图,表明Target浓度在0.02-2nM范围内与荧光强度呈线性关系:y=639.74x+605.44(R
表1 HCR反应序列及CRISPR/Cas12a识别体系序列
实施例2
分别在Target序列的基础上进行单位点突变(MP-SM)、双位点突变(MP-DM)、三位点突变(MP-TM)、四位点突变(MP-FM),所得到的突变序列与牛痘病毒特征核酸片段(Cowpoxvirus,CPXV)、天花病毒特征核酸片段(Variola virus,VARV)、非洲猪瘟病毒特征核酸片段(ASFV)和弓形虫特征核酸片段(Toxoplasma Gondii,TOXO)的序列如表2所示。采用实施例1的制备方法,将Target替换成各核酸片段,使用同样的检测方法和条件进行荧光检测,检测结果如图3所示,表明该体系对猴痘病毒的特征核酸具有高特异性。
表2选择性实验序列
实施例3
基于DNA-铜纳米簇与HCR信号放大的CRISPR/Cas12a报告技术检测猴痘病毒特征核酸片段质粒
(1)制备PCR Mix:将1×Taq Master Mix、0.4μM F-Primer、0.4μM R-Primer、ddH
(2)PCR反应:将2μL不同浓度猴痘病毒特征核酸质粒(2.5copies/μL、5copies/μL、10copies/μL、10
(3)采用实施例1的方法,将Target替换成0.2μL不同浓度质粒的PCR扩增产物,使用同样的检测方法和条件进行荧光检测,检测结果如图4所示,浓度为5copies/μL的猴痘病毒质粒即可引起反应体系荧光显著增强,说明其检测限达到5copies/μL。
实施例4
采用实施例3的制备方法,以猴痘病毒特征核酸质粒(MPXV,GenBank_ON568298_ON563414)、牛痘病毒特征核酸质粒(CPXV,GenBank_X75158)、天花病毒特征核酸质粒(VARV,GenBank_AY552594)、非洲猪瘟病毒特征核酸质粒(ASFV,GenBank_MN809121)和弓形虫特征核酸质粒(TOXO,GenBank_AF146527)为检测对象,使用同样的检测方法和条件进行荧光检测,其中MPXV的检测浓度为10
表3选择性质粒及引物序列
- 一种基于DNA-银纳米簇分子信标与核酸外切酶III循环信号放大的转录因子检测方法
- 一种基于Y型DNA模板荧光铜纳米簇的核酸信号放大检测试剂盒及检测方法
